TwoSampleMR: Менделевский анализ (2)
Опять практика кода~
Пример 1. В качестве примера взят ИМТ и ишемическая болезнь сердца.
Загрузить пакет
library(TwoSampleMR)
library(ggplot2)
Получить данные о воздействии и результатах
bmi_exp_dat <- extract_instruments(outcomes = 'ieu-a-2')
chd_out_dat <- extract_outcome_data(snps = bmi_exp_dat$SNP, outcomes = 'ieu-a-7')
extract_instrumentsПараметры функции следующие::
extract_instruments(
outcomes,
p1 = 5e-08,
clump = TRUE,
p2 = 5e-08,
r2 = 0.001,
kb = 10000,
access_token = ieugwasr::check_access_token(),
force_server = FALSE
)
Вы можете настроить значения p, r2 и kb в соответствии со своими потребностями.
Значение F и значение R^2
Что касается значения F и значение R^2расчет,Я уже упомянул все шаги по воспроизведению МР-анализа сегодня для системы.,Давайте еще раз запишем:
Пучокhyperten_liberalЗамените его своим собственным воздействием.данные Вот и все
# Calculate R2 and F stat for exposure data
# Liberal hypertension F stat
hyperten_liberal$r2 <- (2 * (hyperten_liberal$beta.exposure^2) * hyperten_liberal$eaf.exposure * (1 - hyperten_liberal$eaf.exposure)) /
(2 * (hyperten_liberal$beta.exposure^2) * hyperten_liberal$eaf.exposure * (1 - hyperten_liberal$eaf.exposure) +
2 * hyperten_liberal$N * hyperten_liberal$eaf.exposure * (1 - hyperten_liberal$eaf.exposure) * hyperten_liberal$se.exposure^2)
hyperten_liberal$F <- hyperten_liberal$r2 * (hyperten_liberal$N - 2) / (1 - hyperten_liberal$r2)
hyperten_liberal_meanF <- mean(hyperten_liberal$F)
# Stringent hypertension F stat
hyperten_stringent$r2 <- (2 * (hyperten_stringent$beta.exposure^2) * hyperten_stringent$eaf.exposure * (1 - hyperten_stringent$eaf.exposure)) /
(2 * (hyperten_stringent$beta.exposure^2) * hyperten_stringent$eaf.exposure * (1 - hyperten_stringent$eaf.exposure) +
2 * hyperten_stringent$N * hyperten_stringent$eaf.exposure * (1 - hyperten_stringent$eaf.exposure) * hyperten_stringent$se.exposure^2)
hyperten_stringent$F <- hyperten_stringent$r2 * (hyperten_stringent$N - 2) / (1 - hyperten_stringent$r2)
hyperten_stringent_meanF <- mean(hyperten_stringent$F)
harmonise
dat <- harmonise_data(bmi_exp_dat, chd_out_dat)
Менделевская рандомизация
После разоблаченияирезультатданныеодеялоharmonisedпосле,У нас есть характеристики воздействия и результата для каждого инструмента. SNP Размер эффекта и стандартная ошибка. Мы можем использовать эту информацию для проведения Менделевской рандомизация Ла~
res <- mr(dat)
res
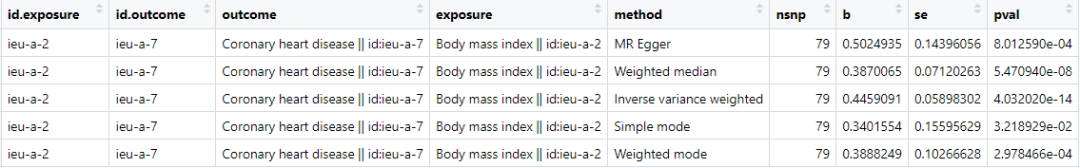
Посмотреть список методов МР
methods <- mr_method_list()
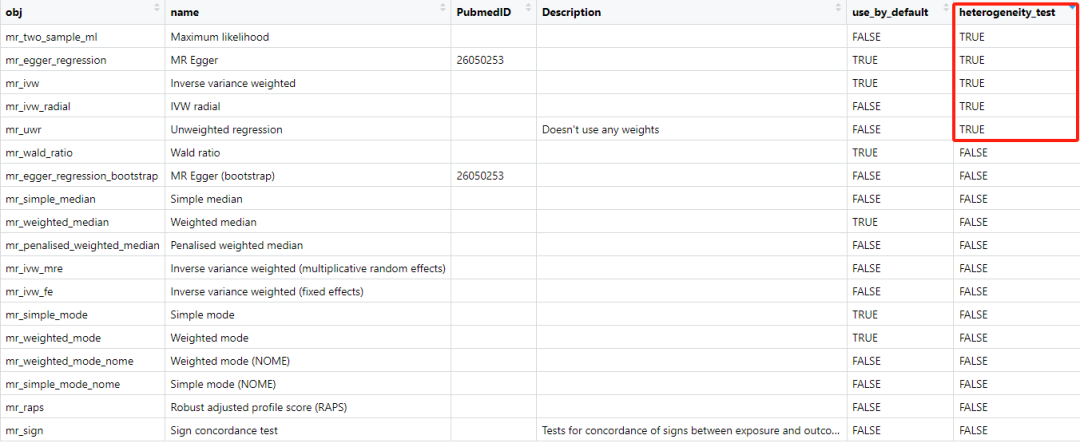
Как видите, существует 5 методов, которые можно использовать для проверки неоднородности.
также,пройтиmrэтот шагпосле,Получаем значения b, seиp,Но если вы хотите нарисовать Карту леса, нам все еще нужно числовое значение доверительного интервала. Что делать?
Генерируйте отношения шансов с 95% доверительными интервалами
generate_odds_ratios(res)
анализ чувствительности
статистика неоднородности
mr_heterogeneity(dat)
Выполните MR Egger и верните значение перехвата.
mr_pleiotropy_test(dat)
Результат возврата следующий:
id.exposure id.outcome outcome
1 ieu-a-2 ieu-a-7 Coronary heart disease || id:ieu-a-7
exposure egger_intercept se pval
1 Body mass index || id:ieu-a-2 -0.001719304 0.003985962 0.6674266
MR Egger Член перехвата в регрессии равен MR анализироватьрезультат Является ли оно целевым?горизонтальная плейотропияПолезные индикаторы воздействия。
Анализ одного SNP
Чтобы получить оценки MR для каждого SNP индивидуально, мы можем сделать следующее:
res_single <- mr_singlesnp(dat)
метаанализ фиксированных эффектов
res_single <- mr_singlesnp(dat, single_method = "mr_meta_fixed")
метод соотношения коэффициентов
res_single <- mr_singlesnp(dat, all_method = "mr_two_sample_ml")
mr_singlesnp()Функция также использует все доступные SNP Вычисление завершено MR, используется по умолчанию IVW и MR Egger метод. Это можно указать следующим образом:
mr_singlesnp(
dat,
parameters = default_parameters(),
single_method = "mr_wald_ratio",
all_method = c("mr_ivw", "mr_egger_regression")
)
Анализ по принципу исключения
res_loo <- mr_leaveoneout(dat)
По умолчанию используется взвешивание обратной дисперсии, но этого можно добиться, используя method Параметры изменены.
Нарисуй картинку
График рассеяния
res <- mr(dat)
p1 <- mr_scatter_plot(res, dat)
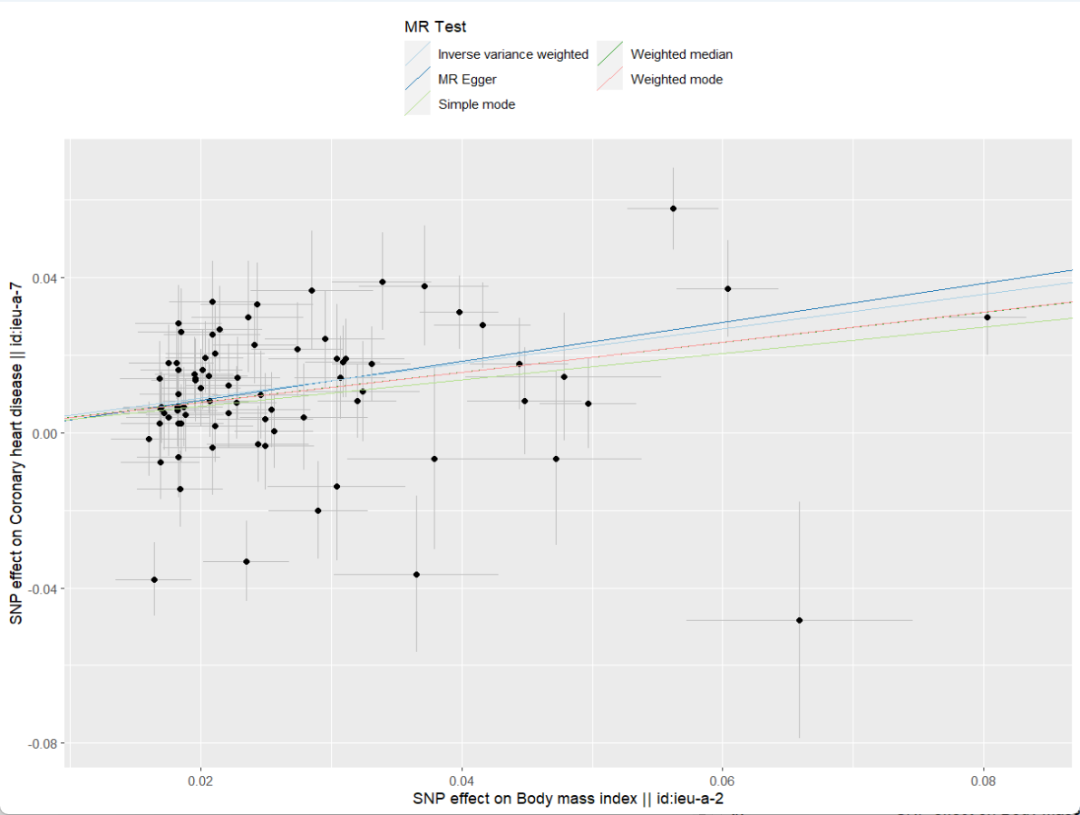
держать:
ggsave(p1[[1]], file = "filename.pdf", width = 7, height = 7)
ggsave(p1[[1]], file = "filename.png", width = 7, height = 7)
Карта леса
res_single <- mr_singlesnp(dat)
p2 <- mr_forest_plot(res_single)
p2[[1]]
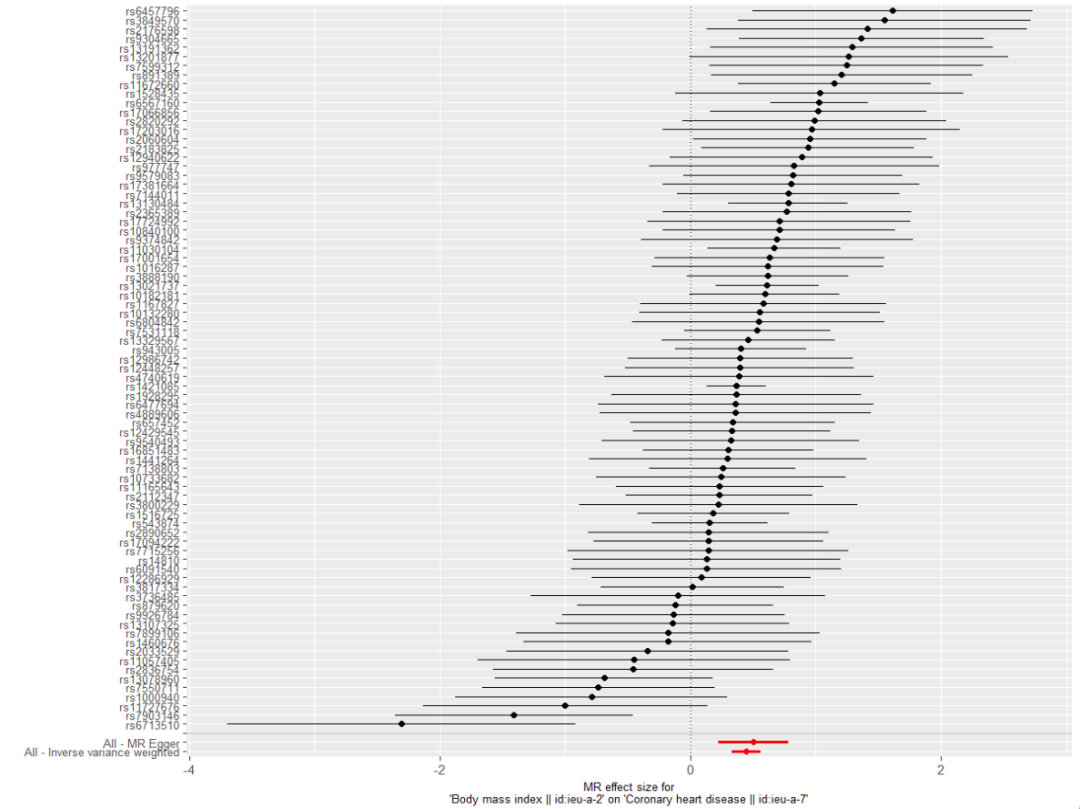
«Оставить один»
res_loo <- mr_leaveoneout(dat)
p3 <- mr_leaveoneout_plot(res_loo)
p3[[1]]
воронкообразная диаграмма
res_single <- mr_singlesnp(dat)
p4 <- mr_funnel_plot(res_single)
p4[[1]]
Пример 2: Несколько экспозиций соответствуют одному финалу.
Получить данные о воздействии и результатах
exp_dat <- extract_instruments(outcomes = c(2, 100, 1032, 104, 1, 72, 999))
table(exp_dat$exposure)
chd_out_dat <- extract_outcome_data(
snps = exp_dat$SNP,
outcomes = 7
)
Номер, соответствующий идентификатору:
2 -> ieu-a-2
100 -> ieu-a-100
1032 -> ieu-a-1032
104 -> ieu-a-104
1 -> ieu-a-1
72 -> ieu-a-72
999 -> ieu-a-999
table(exp_dat$exposure)
Adiponectin || id:ieu-a-1 Body fat || id:ieu-a-999
14 10
Body mass index || id:ieu-a-2 Hip circumference || id:ieu-a-100
79 2
Waist-to-hip ratio || id:ieu-a-72 Waist circumference || id:ieu-a-104
31 1
Если вы используете здесь свои собственные данные,
expdat <- mv_extract_exposures_local(
filenames_exposure,
sep = " ",
phenotype_col = "Phenotype",
snp_col = "SNP",
beta_col = "beta",
se_col = "se",
eaf_col = "eaf",
effect_allele_col = "effect_allele",
other_allele_col = "other_allele",
pval_col = "pval",
units_col = "units",
ncase_col = "ncase",
ncontrol_col = "ncontrol",
samplesize_col = "samplesize",
gene_col = "gene",
id_col = "id",
min_pval = 1e-200,
log_pval = FALSE,
pval_threshold = 5e-08,
clump_r2 = 0.001,
clump_kb = 10000,
harmonise_strictness = 2
)
harmonise&mr
dat2 <- harmonise_data(
exposure_dat = exp_dat,
outcome_dat = chd_out_dat
)
res <- mr(dat2)
Здесь вы можете использовать многомерный MR-анализ следующим образом:
mvdat <- mv_harmonise_data(exposure_dat, outcome_dat)
res <- mv_multiple(mvdat)
Нарисуй картинку
res <- subset_on_method(res) # По умолчанию, на основе IVW метод(>1 индивидуальныйинструмент СНП) или Wald метод соотношений (1 индивидуальныйинструмент SNP) для подмножества.
res <- sort_1_to_many(res, b = "b", sort_action = 4) # Результаты будут отсортированы по уменьшению размера эффекта (эффект в верхней части графика является наибольшим).
res <- split_exposure(res) # чтобы сохранить Y Аккуратность меток осей мы выставим ID Ярлык исключен из столбца воздействия
res$weight <- 1/res$se
min(exp(res$b - 1.96*res$se)) # identify value for 'lo' in forest_plot_1_to_many
max(exp(res$b + 1.96*res$se)) # identify value for 'up' in forest_plot_1_to_many
Карта леса
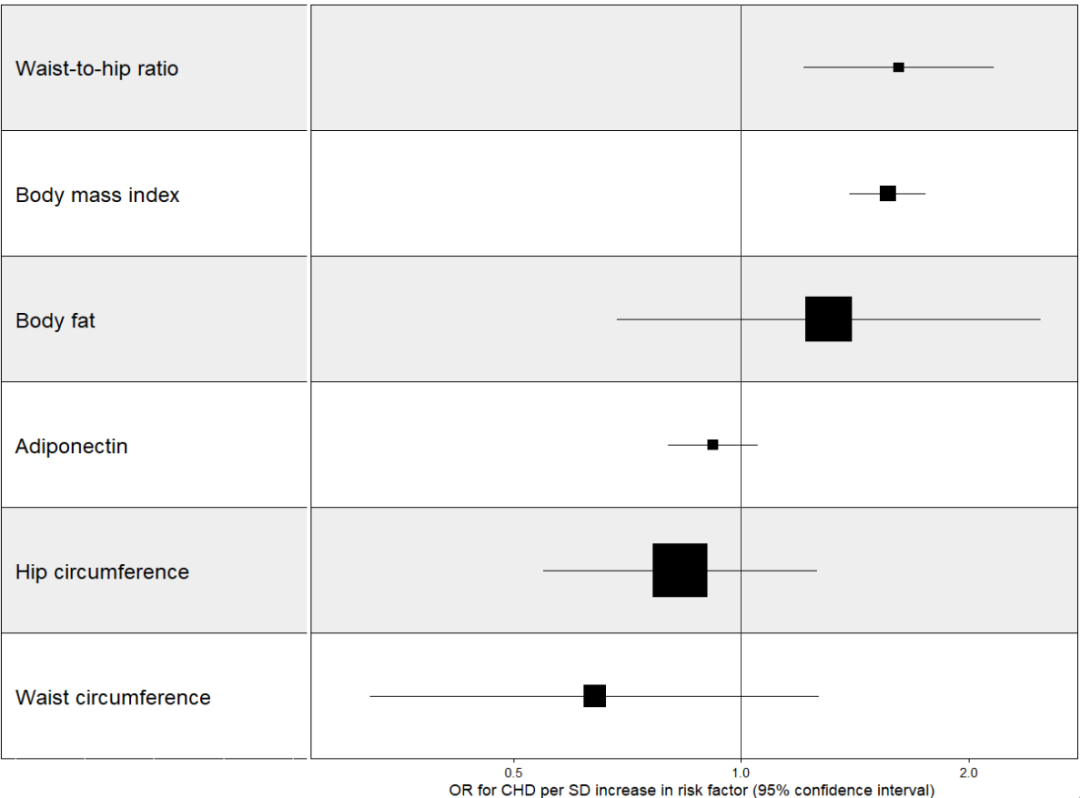
Какая уродливая картина! ! !
Посмотрим, сможем ли мы это улучшить~
res <- mr(dat2)
tmp <- generate_odds_ratios(res)
tmp$` ` <- paste(rep(" ", 22), collapse = " ") ##Создать пустую строку
# Создать ИЛИ (95% CI)
tmp$`OR (95% CI)` <- sprintf("%.2f (%.2f to %.2f)", #sprintF возвращает символ и, который можно комбинировать с переменной
tmp$or,tmp$or_lci95,tmp$or_uci95)
Карта леса Улучшенная версия
p1 <- forest(tmp[,c(4:6,14,13,9)], ##Выберите здесь в соответствии с желаемым контентом
est = tmp$or, #Значение эффекта
lower = tmp$or_lci95, #Нижний предел доверительного интервала
upper = tmp$or_uci95, #верхний предел доверительного интервала
sizes = tmp$se,
ci_column = 5, #drawКарта в этой рубрике леса, выберите пустой столбец
ref_line = 1,
arrow_lab = c("No CHD", "CHD"),
xlim = c(0, 4),
ticks_at = c(0.5, 1, 2, 3),
footnote = "")
p1 ##Выглядит неплохо
Сохранить изображение:
require(ggplotify)
p2 <- as.ggplot(p2)
class(p2)
ggsave(p2,file="forest.pdf",width = 10,height = 10)
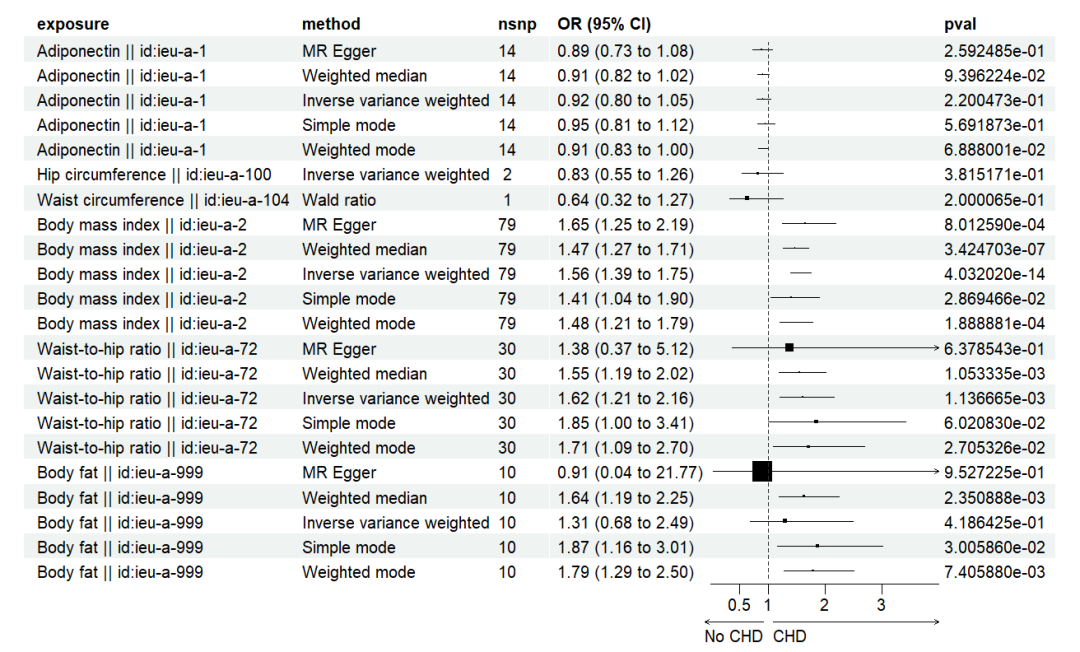
Пример 3: Одному воздействию соответствует несколько концовок.
Получить данные о воздействии и результатах
exp_dat <- extract_instruments(outcomes = 2) # extract instruments for BMI
ao <- available_outcomes()
ao <- ao[ao$category == "Disease", ] # identify diseases
ao <- ao[which(ao$ncase > 100), ]
dis_dat <- extract_outcome_data(
snps = exp_dat$SNP,
outcomes = ao$id
)
dat3 <- harmonise_data(
exposure_dat = exp_dat,
outcome_dat = dis_dat
)
res <- mr(dat3, method_list = c("mr_wald_ratio", "mr_ivw"))
res <- split_outcome(res)
Дополнительные идеи для рисования см. в примере 2~.
Бесшовная интеграция с пакетом MendelianRandomization.
# Convert to the `MRInput` format for the MendelianRandomization package:
dat2 <- dat_to_MRInput(dat)
# This produces a list of `MRInput` objects that can be used with the MendelianRandomization functions, e.g.
MendelianRandomization::mr_ivw(dat2[[1]])
# Alternatively, convert to the `MRInput` format but also obtaining the LD matrix for the instruments
dat2 <- dat_to_MRInput(dat, get_correlation = TRUE)
MendelianRandomization::mr_ivw(dat2[[1]], correl = TRUE)
MR-MoE: Метод машинного обучения
# Extact instruments for BMI
exposure_dat <- extract_instruments("ieu-a-2")
# Get corresponding effects for CHD
outcome_dat <- extract_outcome_data(exposure_dat$SNP, "ieu-a-7")
# Harmonise
dat <- harmonise_data(exposure_dat, outcome_dat)
# Load the downloaded RData object. This loads the rf object
load("rf.rdata") ##Эти данные Скачать➡dropbox.com/s/5la7y38od95swcf
# Obtain estimates from all methods, and generate data metrics
res <- mr_wrapper(dat)
# MR-MoE - predict the performance of each method
res_moe <- mr_moe(res, rf)
Описание параметра:
- `estimates` (results from each MR)
- `heterogeneity` (results from heterogeneity for different filtering approaches)
- `directional_pleiotropy` (egger intercepts)
- `info` (metrics used to generate MOE)

Углубленный анализ переполнения памяти CUDA: OutOfMemoryError: CUDA не хватает памяти. Попыталась выделить 3,21 Ги Б (GPU 0; всего 8,00 Ги Б).
[Решено] ошибка установки conda. Среда решения: не удалось выполнить первоначальное зависание. Повторная попытка с помощью файла (графическое руководство).
Прочитайте нейросетевую модель Трансформера в одной статье

.ART Теплые зимние предложения уже открыты

Сравнительная таблица описания кодов ошибок Amap

Уведомление о последних правилах Points Mall в декабре 2022 года.

Даже новички могут быстро приступить к работе с легким сервером приложений.
Взгляд на RSAC 2024|Защита конфиденциальности в эпоху больших моделей
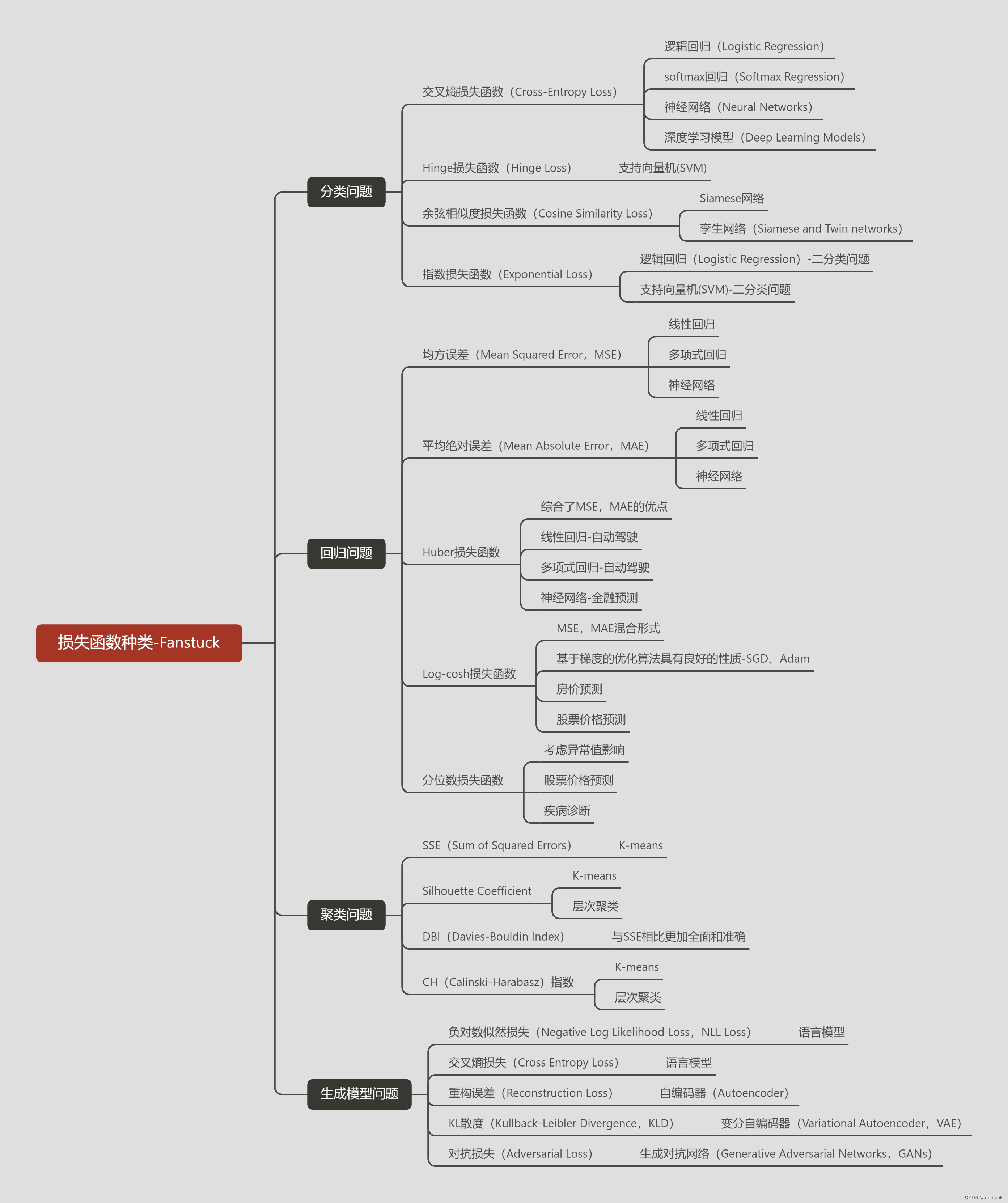
Вы используете ИИ каждый день и до сих пор не знаете, как ИИ дает обратную связь? Одна статья для понимания реализации в коде Python общих функций потерь генеративных моделей + анализ принципов расчета.

Используйте (внутренний) почтовый ящик для образовательных учреждений, чтобы использовать Microsoft Family Bucket (1T дискового пространства на одном диске и версию Office 365 для образовательных учреждений)
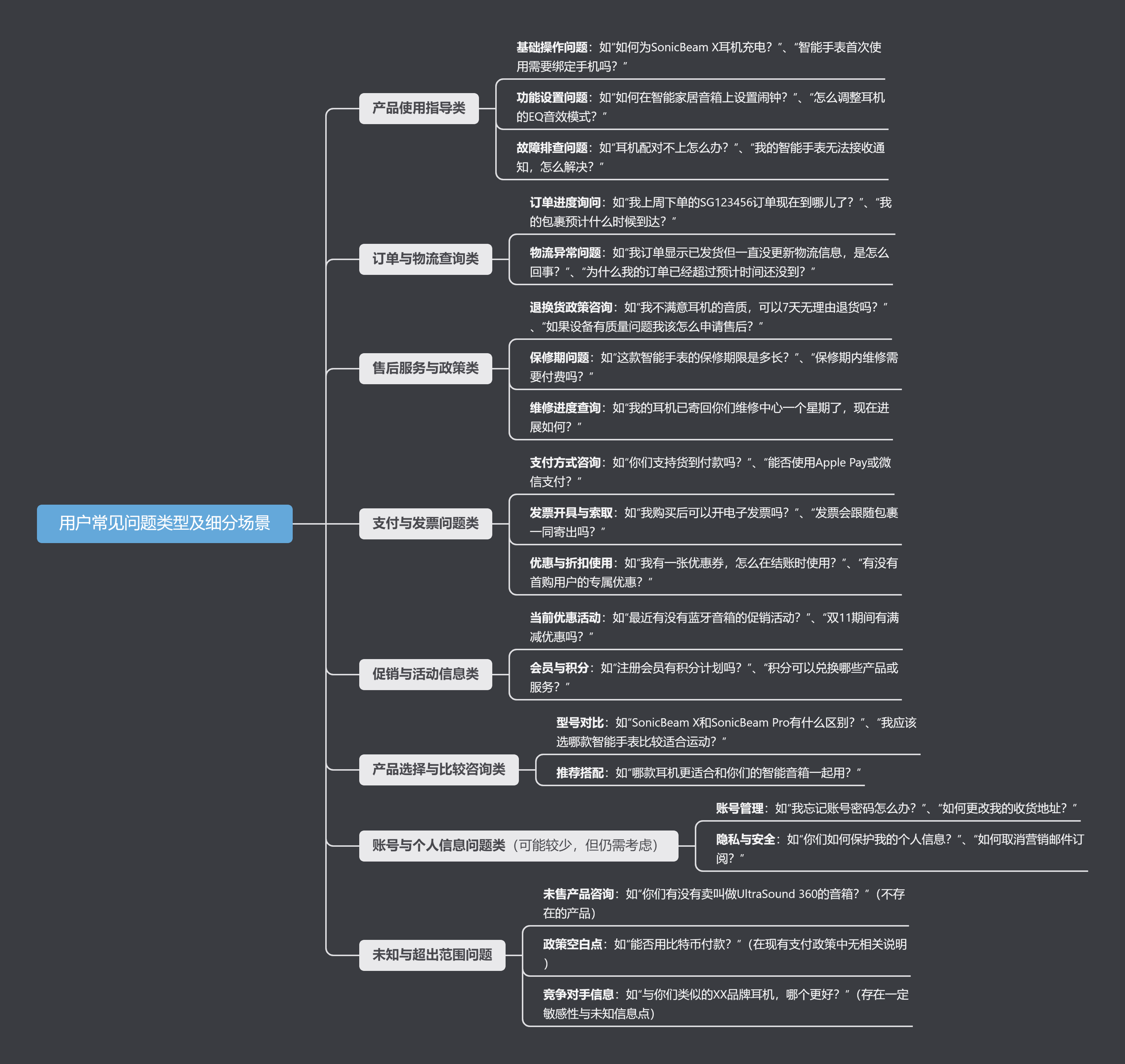
Руководство по началу работы с оперативным проектом (7) Практическое сочетание оперативного письма — оперативного письма на основе интеллектуальной системы вопросов и ответов службы поддержки клиентов

[docker] Версия сервера «Чтение 3» — создайте свою собственную программу чтения веб-текста

Обзор Cloud-init и этапы создания в рамках PVE
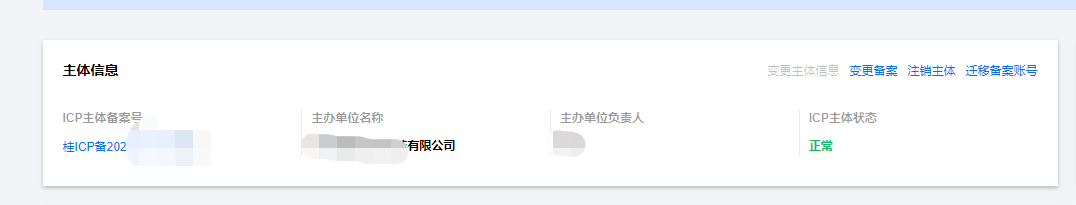
Корпоративные пользователи используют пакет регистрационных ресурсов для регистрации ICP для веб-сайта и активации оплаты WeChat H5 (с кодом платежного узла версии API V3)

Подробное объяснение таких показателей производительности с высоким уровнем параллелизма, как QPS, TPS, RT и пропускная способность.
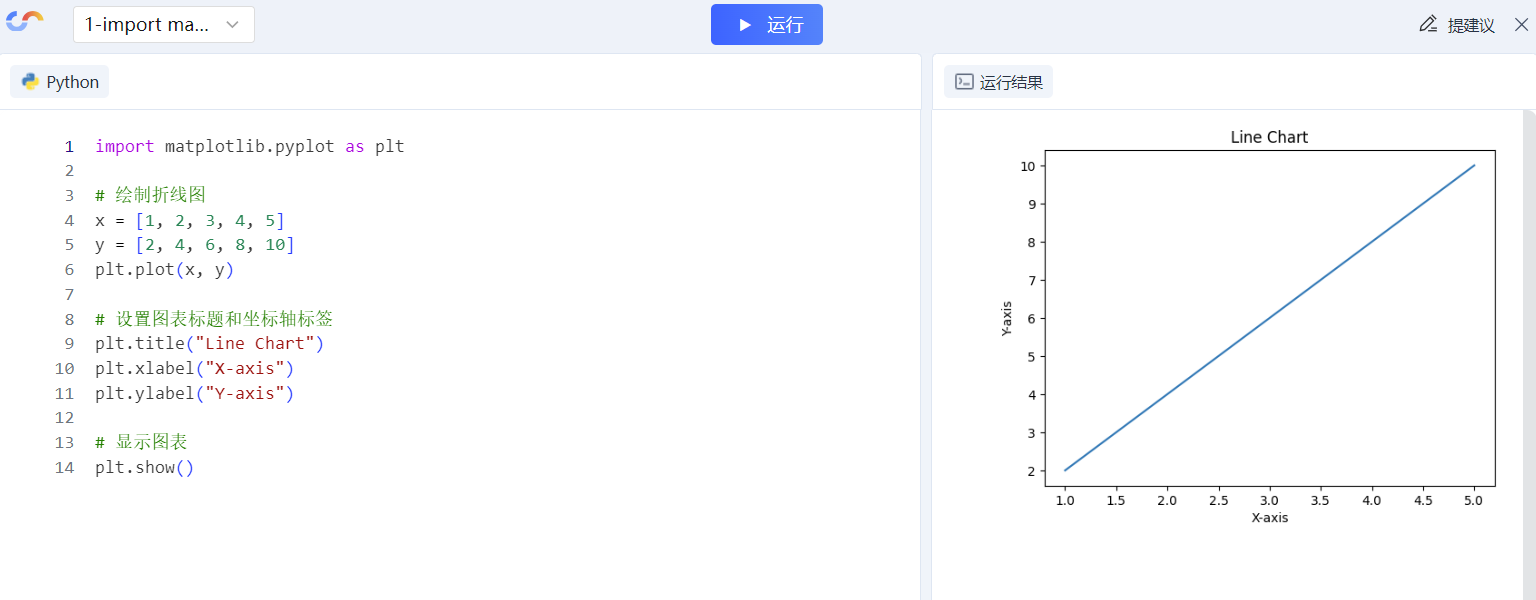
Удачи в конкурсе Python Essay Challenge, станьте первым, кто испытает новую функцию сообщества [Запускать блоки кода онлайн] и выиграйте множество изысканных подарков!

[Техническая посадка травы] Кровавая рвота и отделка позволяют вам необычным образом ощипывать гусиные перья! Не распространяйте информацию! ! !

[Официальное ограниченное по времени мероприятие] Сейчас ноябрь, напишите и получите приз

Прочтите это в одной статье: Учебник для няни по созданию сервера Huanshou Parlu на базе CVM-сервера.

Cloud Native | Что такое CRD (настраиваемые определения ресурсов) в K8s?

Как использовать Cloudflare CDN для настройки узла (CF самостоятельно выбирает IP) Гонконг, Китай/Азия узел/сводка и рекомендации внутреннего высокоскоростного IP-сегмента

Дополнительные правила вознаграждения амбассадоров акции в марте 2023 г.
Можно ли открыть частный сервер Phantom Beast Palu одним щелчком мыши? Супер простой урок для начинающих! (Прилагается метод обновления сервера)
[Играйте с Phantom Beast Palu] Обновите игровой сервер Phantom Beast Pallu одним щелчком мыши

Maotouhu делится: последний доступный внутри страны адрес склада исходного образа Docker 2024 года (обновлено 1 декабря)

Кодирование Base64 в MultipartFile
5 точек расширения SpringBoot, супер практично!

Глубокое понимание сопоставления индексов Elasticsearch.