Оценка программного обеспечения: преобразование объектов Anndata и Seurat миллионов одноячеечных данных
Недавно я, наконец, закончил предзащиту докторской и отправлю ее на слепое рецензирование после капитального ремонта. Пожелайте мне удачи~.
С бурным ростом результатов исследований, связанных с отдельными клетками, область одноклеточных клеток вступила в эпоху миллионов или даже десятков миллионов клеток. Поэтому многие энтузиасты языка R (включая меня) начали изучать Python и использовать процесс Scanpy. Однако, поскольку я привык к процессу Сера, иногда мне нужно преобразовать данные одной ячейки объектов Anndata в объекты Сера, а затем использовать язык R для выполнения некоторого анализа. Самая большая проблема заключается в том, как плавно преобразовать формат h5ad объектов Anndata в объекты Seurat. В этой статье оцениваются и обобщаются различные программы взаимного преобразования на основе данных испытаний отдельных ячеек на миллионном уровне. Надеюсь, это поможет всем ~
1. Данные испытаний
Прежде всего, данные испытаний взяты из статьи «Single-cell RNA-seq выявляет молекулярные и генетические связи типа клеток с волчанкой» о секвенировании одноклеточных PBMC системной красной волчанки. Данные хранятся в базе данных GEO GSE174188, формат хранения — h5ad, и содержит около 1,2 миллиона отдельных ячеек. После нажатия кнопки «Загрузить» мы начали оценивать программное обеспечение для взаимной конвертации.
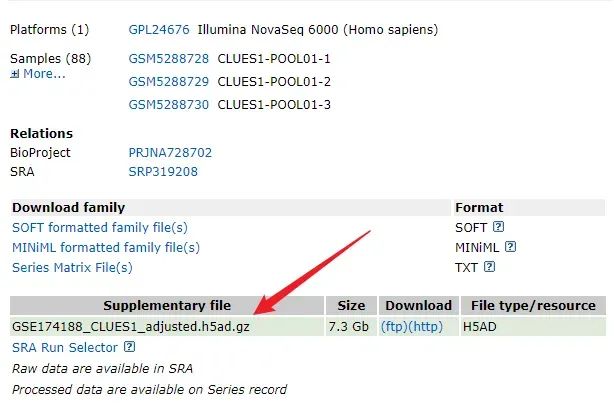
image-20240321174743755
Загруженные данные h5ad распаковываются и получают имя GSE174188_CLUES1_adjusted.h5ad, размер которого составляет примерно 10,6 ГБ.
2. Оценка взаимной передачи
Я собрал 4 метода преобразования формата h5ad объектов nndata и объектов Seurat, в том числе
- Пакет R SeuratDisk,Это алгоритм сопоставления Сёра (если он прост в использовании),Нам не нужно беспокоиться о других пакетах.,Так что вполне возможно,Этот алгоритм определенно не прост в использовании...);
- пакет R;
- Пакет R MuDataSeurat;
- Пакет R и пакет Python scDIOR,Этот алгоритм часто используется в моих предыдущих публикациях и настоятельно рекомендуется, но развертывание среды вызывает некоторые затруднения.
Давайте оценим по порядку:
1. Пакет R SeuratDisk
library(Seurat)
library(SeuratDisk)
# 1.1 h5ad к Сёра
time.py2R = system.time({
Convert('./Rawdata/GSE174188_CLUES1_adjusted.h5ad', "h5seurat",
overwrite = TRUE,assay = "RNA")
})
print(time.py2R)
time.read = system.time({
seurat_obj <- LoadH5Seurat("./Rawdata/GSE174188_CLUES1_adjusted.h5ad")
})
print(time.read)
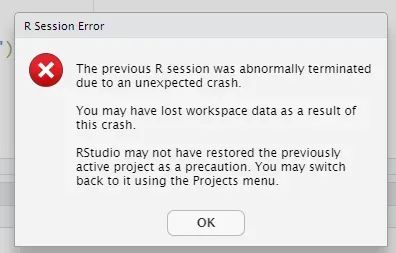
image-20240321181135420
Операция преобразования на языке R завершилась сбоем, и никаких результатов не было...
Здесь снова попробуйте конвертировать Seurat в h5ad:
seurat_object = read_rds("./Outdata/Step1.seurat.data.Clean.rds")
# 1.2 Сёра в h5ad
time.R2py = system.time({
seu = DietSeurat(
seurat_object,
counts = TRUE, # so, raw counts save to adata.raw.X
data = TRUE, # so, log1p counts save to adata.X
scale.data = FALSE, # set to false, or else will save to adata.X
features = rownames(seurat_object), # export all genes, not just top highly variable genes
assays = "RNA",
dimreducs = c("pca","umap"),
graphs = c("RNA_nn", "RNA_snn"), # to RNA_nn -> distances, RNA_snn -> connectivities
misc = TRUE
)
# step 2: factor to character, or else your factor will be number in adata
i <- sapply(seu@meta.data, is.factor)
seu@meta.data[i] <- lapply(seu@meta.data[i], as.character)
# step 3: convert
SaveH5Seurat(seu, filename = "./Outdata/SeuratDisk.h5seurat", overwrite = TRUE)
Convert("./Outdata/SeuratDisk.h5seurat", "./Outdata/SeuratDisk.h5ad", assay="RNA", overwrite = TRUE)
})
print(time.R2py)
user system elapsed 577.649 32.573 611.104
При использовании пакета SeuratDisk для преобразования Seurat в h5ad преобразование одного миллиона отдельных ячеек заняло более 600 секунд, что относительно медленно.
2. Пакет R прост
Установка пакета R немного затруднительна. Сначала вам нужно установить некоторое программное обеспечение под Linux:
conda install anndata==0.6.19 scipy==1.2.1 -c bioconda
conda install loompy -c biocond
Затем установите его на языке R:
devtools::install_github("cellgeni/sceasy")
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(c("LoomExperiment", "SingleCellExperiment"))
install.packages('reticulate')
Затем вы можете запустить sceasy:
library(sceasy)
library(reticulate)
library(Seurat)
# 2.1 h5ad к Сёра
time.py2R = system.time({
sceasy::convertFormat(obj = "./Rawdata/GSE174188_CLUES1_adjusted.h5ad", from="anndata",
to="seurat",outFile = 'sceasy.rds')
})
print(time.py2R)
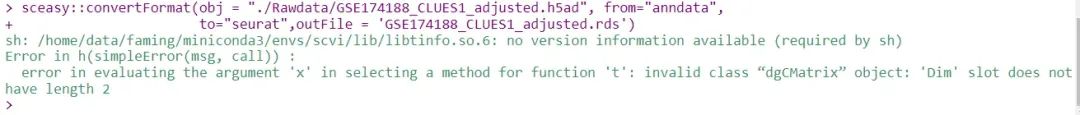
image-20240321194652273
Кажется, что объем одной ячейки слишком велик, и сообщается об ошибке.
Попробуйте конвертировать Seurat в h5ad:
# 2.2 Сёра в h5ad
time.R2py = system.time({
sceasy::convertFormat(seurat_object, from="seurat", to="anndata",
outFile='./Outdata/sceasy.h5ad')
})
print(time.R2py)
user system elapsed 243.205 22.359 267.202
Используя пакет sceasy для преобразования Seurat в h5ad, преобразование одного миллиона отдельных ячеек заняло более 267 секунд, что относительно быстро.
3. R пакет MuDataSeurat
Пакет R MuDataSeurat относительно прост в установке:
remotes::install_github("zqfang/MuDataSeurat", ref='dev', force = T)
Затем запустите преобразование:
library(MuDataSeurat)
# 3.1 h5ad к Сёра
time.py2R = system.time({
seurat.data = MuDataSeurat::ReadH5AD(file = "./Outdata/GSE174188_CLUES1_adjusted.h5ad")
})
print(time.py2R)

image-20240321202345088
Сообщили об ошибке.
Попробуйте конвертировать Seurat в h5ad:
# 3.2 Сёра в h5ad
time.R2py = system.time({
MuDataSeurat::WriteH5AD(seurat_object, "MuDataSeurat.h5ad", assay="RNA")
})
print(time.R2py)
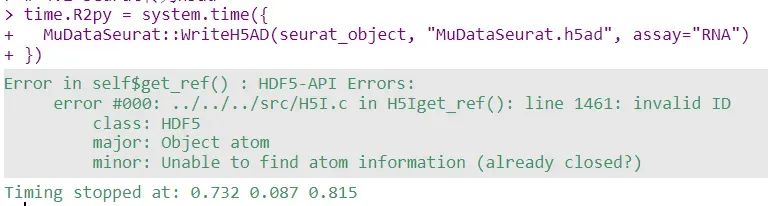
image-20240321202804306
Все еще сообщает об ошибке.
4. Пакет R dior и пакет Python scDIOR.
Этот алгоритм часто используется в моих предыдущих постах. Его сложно установить, поскольку зависимые пакеты имеют ограничения по версии, иначе будет сообщено об ошибке. Поэтому я предлагаю создать небольшую среду Conda для scdiopy:
conda create -n scdiopy python==3.8
conda activate scdiopy
# for python
pip install diopy
pip install scanpy==1.9.6 numpy==1.21.3 numba==0.57.1
Затем установите его на языке R:
devtools::install_github('JiekaiLab/dior')
Затем упорядочите преобразование в R:
library(Seurat)
library(dior)
# 4.1 h5ad к Сёра
time.py2R = system.time({
seurat.data <- read_h5ad(file = './Rawdata/GSE174188_CLUES1_adjusted.h5ad',
assay_name = 'RNA',
target.object = 'seurat')
})
print(time.py2R)
user system elapsed 239.829 82.604 322.756
Преобразование h5ad в Seurat заняло более 322 секунд.
# 4.2 Сёра в h5ad
time.R2py = system.time({
sceasy::convertFormat(seurat_object, from="seurat", to="anndata",
outFile='./Outdata/scDIOR.h5ad')
})
print(time.R2py)
user system elapsed 243.635 19.612 262.997
Сёра потребовалось более 262 секунд, чтобы конвертировать h5ad.
3. Резюме
Если вам нужно преобразовать данные Seurat и Anndata/h5ad между миллионами одноячеечных данных, я настоятельно рекомендую использовать пакет R dior и пакет Python scDIOR. Преимуществами являются высокая скорость работы и сильная совместимость данных. Недостатком является зависимость пакета. имеет ограничения по версии, иначе легко сообщить об ошибках.
Во-вторых, для преобразования данных Seurat в h5ad пакеты R dior и R sceasy являются хорошим выбором и являются относительно быстрыми, в то время как пакет R SeuratDisk относительно медленный и не рекомендуется.

Углубленный анализ переполнения памяти CUDA: OutOfMemoryError: CUDA не хватает памяти. Попыталась выделить 3,21 Ги Б (GPU 0; всего 8,00 Ги Б).
[Решено] ошибка установки conda. Среда решения: не удалось выполнить первоначальное зависание. Повторная попытка с помощью файла (графическое руководство).
Прочитайте нейросетевую модель Трансформера в одной статье

.ART Теплые зимние предложения уже открыты

Сравнительная таблица описания кодов ошибок Amap

Уведомление о последних правилах Points Mall в декабре 2022 года.

Даже новички могут быстро приступить к работе с легким сервером приложений.
Взгляд на RSAC 2024|Защита конфиденциальности в эпоху больших моделей
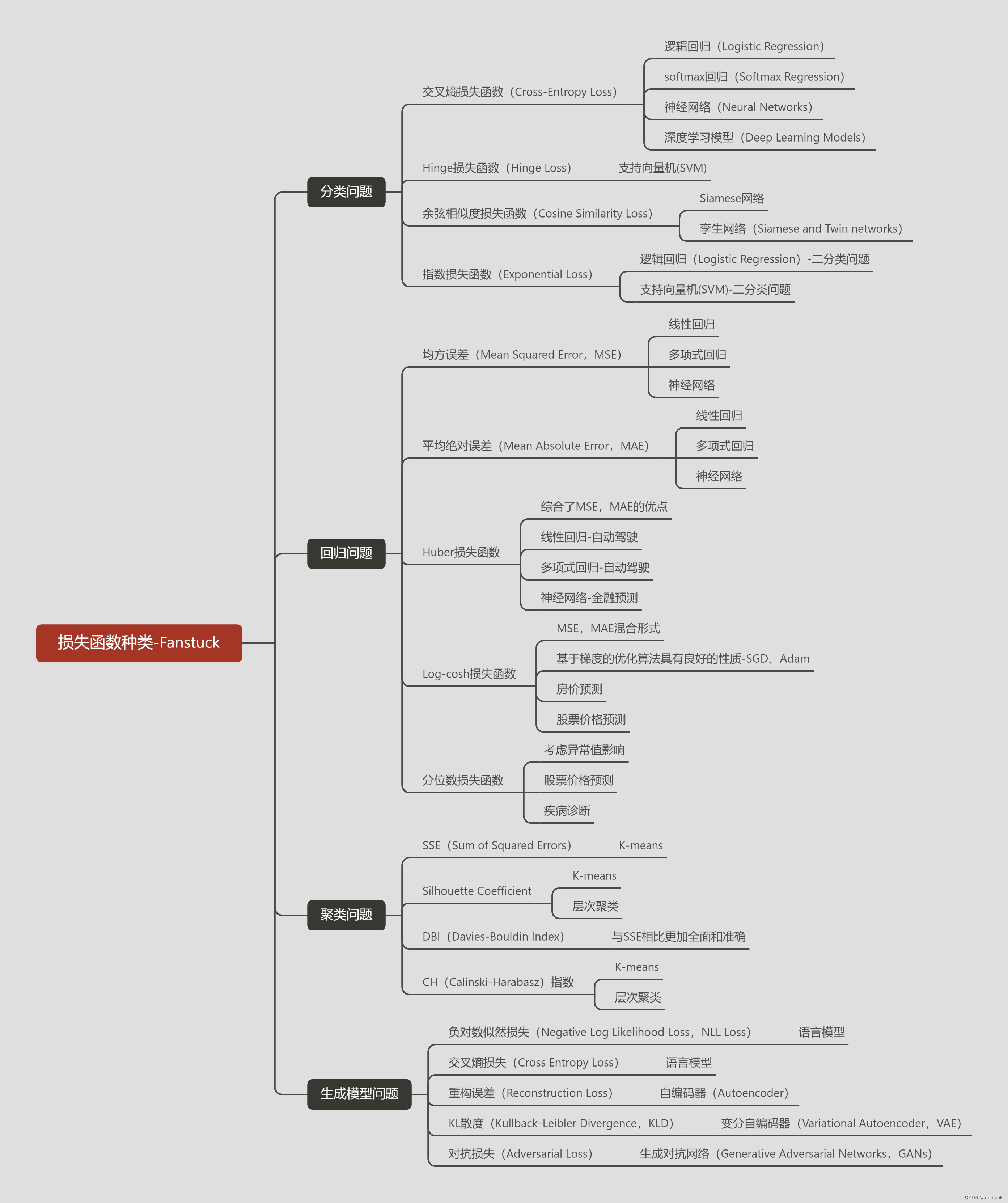
Вы используете ИИ каждый день и до сих пор не знаете, как ИИ дает обратную связь? Одна статья для понимания реализации в коде Python общих функций потерь генеративных моделей + анализ принципов расчета.

Используйте (внутренний) почтовый ящик для образовательных учреждений, чтобы использовать Microsoft Family Bucket (1T дискового пространства на одном диске и версию Office 365 для образовательных учреждений)
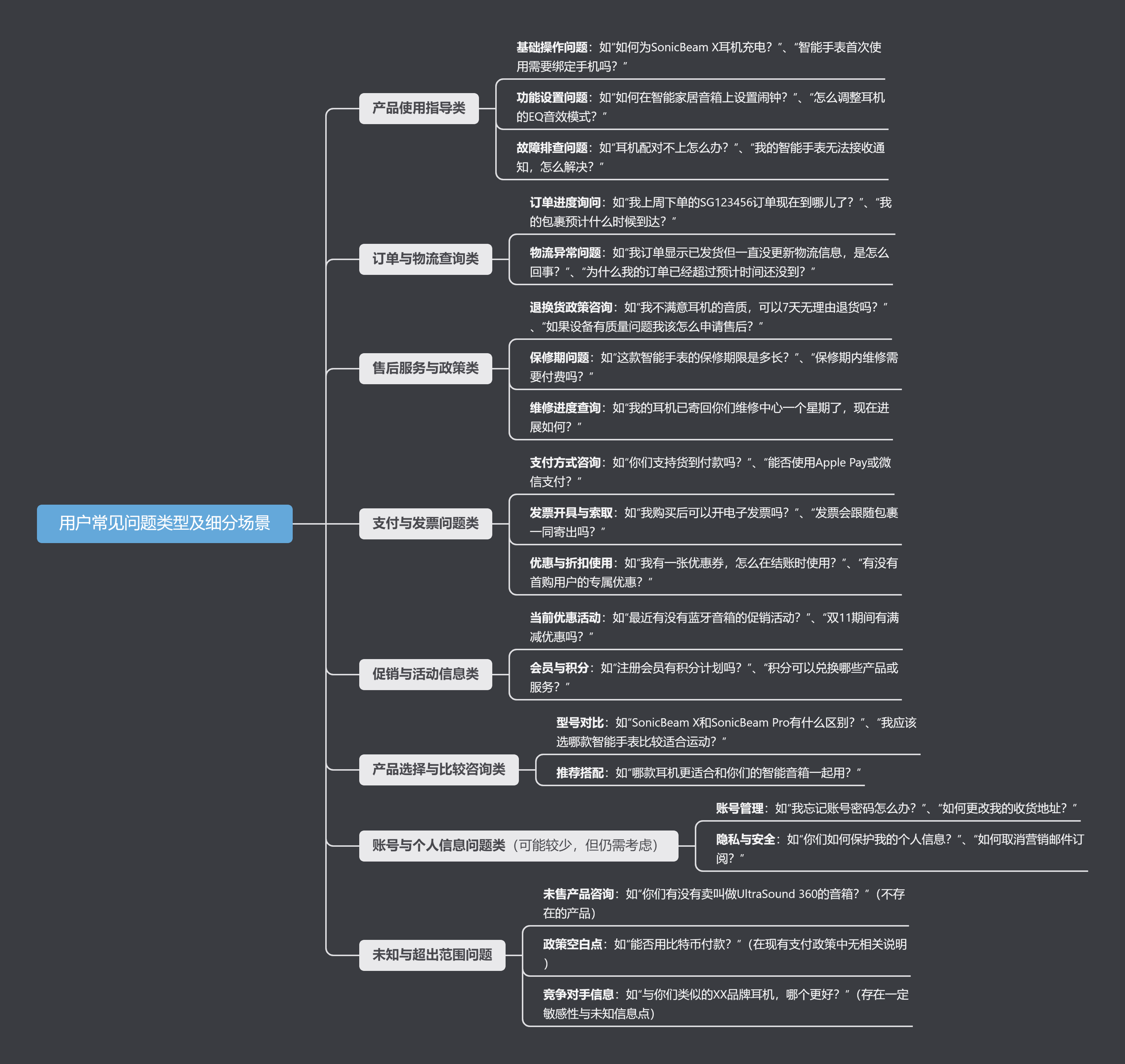
Руководство по началу работы с оперативным проектом (7) Практическое сочетание оперативного письма — оперативного письма на основе интеллектуальной системы вопросов и ответов службы поддержки клиентов

[docker] Версия сервера «Чтение 3» — создайте свою собственную программу чтения веб-текста

Обзор Cloud-init и этапы создания в рамках PVE
Корпоративные пользователи используют пакет регистрационных ресурсов для регистрации ICP для веб-сайта и активации оплаты WeChat H5 (с кодом платежного узла версии API V3)

Подробное объяснение таких показателей производительности с высоким уровнем параллелизма, как QPS, TPS, RT и пропускная способность.
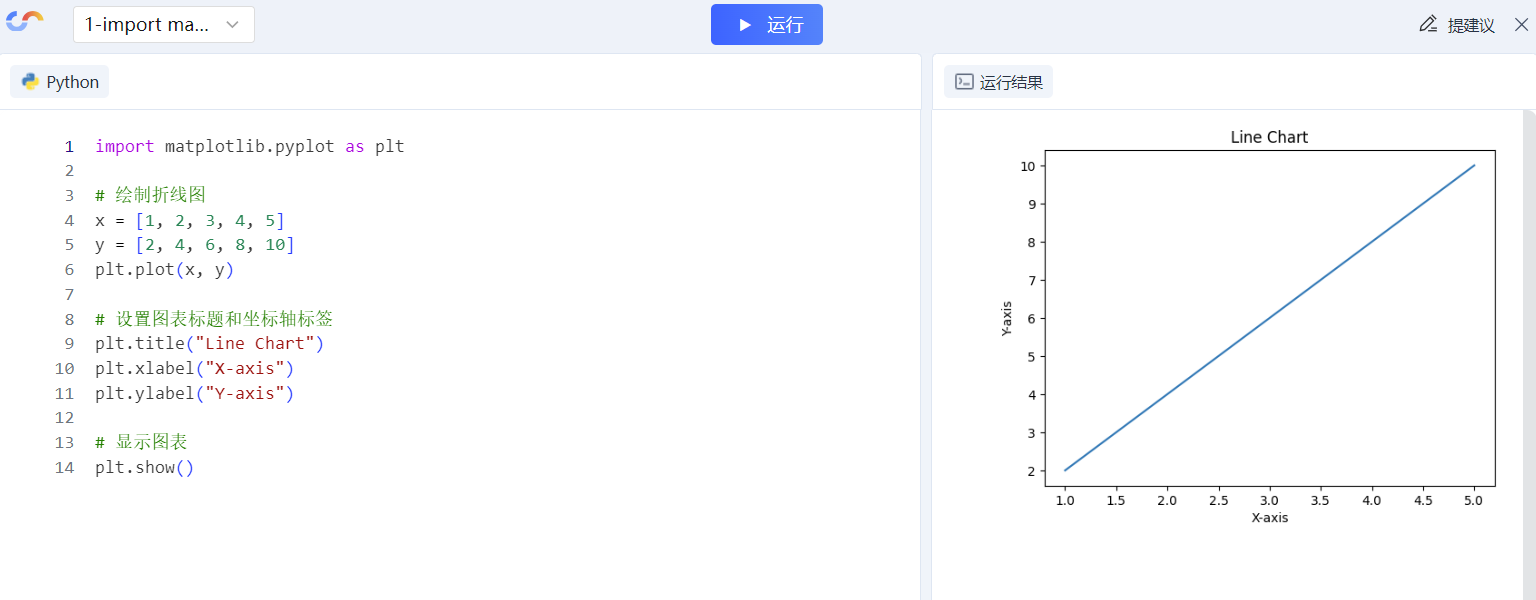
Удачи в конкурсе Python Essay Challenge, станьте первым, кто испытает новую функцию сообщества [Запускать блоки кода онлайн] и выиграйте множество изысканных подарков!

[Техническая посадка травы] Кровавая рвота и отделка позволяют вам необычным образом ощипывать гусиные перья! Не распространяйте информацию! ! !

[Официальное ограниченное по времени мероприятие] Сейчас ноябрь, напишите и получите приз

Прочтите это в одной статье: Учебник для няни по созданию сервера Huanshou Parlu на базе CVM-сервера.

Cloud Native | Что такое CRD (настраиваемые определения ресурсов) в K8s?

Как использовать Cloudflare CDN для настройки узла (CF самостоятельно выбирает IP) Гонконг, Китай/Азия узел/сводка и рекомендации внутреннего высокоскоростного IP-сегмента

Дополнительные правила вознаграждения амбассадоров акции в марте 2023 г.
Можно ли открыть частный сервер Phantom Beast Palu одним щелчком мыши? Супер простой урок для начинающих! (Прилагается метод обновления сервера)
[Играйте с Phantom Beast Palu] Обновите игровой сервер Phantom Beast Pallu одним щелчком мыши

Maotouhu делится: последний доступный внутри страны адрес склада исходного образа Docker 2024 года (обновлено 1 декабря)

Кодирование Base64 в MultipartFile
5 точек расширения SpringBoot, супер практично!

Глубокое понимание сопоставления индексов Elasticsearch.