Обучение и организация анализа сотовой связи с использованием одной выборки Cellchat (V2)
Анализ клеточной коммуникации — это метод анализа, который изучает, как различные типы клеток взаимодействуют и регулируют друг друга посредством сигнальных молекул (таких как лиганды и рецепторы).。Он раскрывает механизмы межклеточных взаимодействий.,Важно понимать, как ткани и органы работают вместе.
Основное содержание анализа сотовой связи следующее:
- Лиганд-рецепторные взаимодействия: Каждый тип клеток экспрессирует определенные лигандыирецептор。Анализ клеточной связи путем изучения этих лигандовирецепторвзаимодействие между,Приходите узнать, как клетки «общаются». Например,Лиганды, выделяемые одной клеткой, могут связываться с рецепторами другой клетки.,Это запускает серию внутриклеточных сигнальных событий.
- активность сигнального пути: Анализируя, какие сигнальные пути активируются в разных типах клеток, исследователи могут узнать, какие сигнальные пути играют роль в конкретных биологических процессах, таких как дифференцировка клеток, иммунные реакции или болезненные состояния.
- Построение межклеточной сети: Анализ сотовой связи обычно строит сетевую диаграмму между ячейками.,Покажите, как разные типы клеток взаимодействуют друг с другом. Эта сеть может помочь идентифицировать основных отправителей и получателей сигналов.,и какие клетки играют доминирующую роль в ткани.
- Количественный анализ: Рассчитав силу клеточной связи (например, вероятность связывания лиганда с рецептором), можно получить Количественный анализ Интенсивность связи между разными типами клеток. Это может показать, какой тип клеток доминирует в общении в конкретных условиях.
- Анализ функционального обогащения: Анализ сотовой связи также может функционального обогащения используются в сочетании для изучения того, какие биологические функции или пути регулируются конкретными клеточными коммуникациями.
Часто используемые инструменты:
1. CellChat. Это широко используемый пакет R, который может идентифицировать и количественно оценивать связь между различными типами ячеек и предоставляет богатые инструменты визуализации.
2. CellPhoneDB: еще один широко используемый инструмент, предназначенный для анализа клеточных коммуникаций на основе взаимодействия лиганд-рецептор.
На этот раз мы в основном узнаем об инструменте Cellchat.
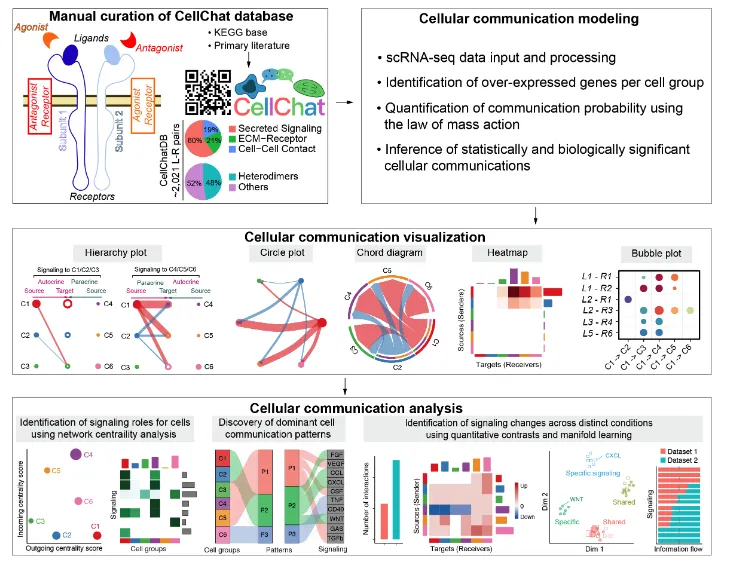

CellChatDB База данных v2 содержит богатую априорную информацию:
1. Содержание базы данных основано на информации о взаимодействии лиганд-рецептор человека и мыши из известной литературы.
2. Эта информация содержит около 3300 подтвержденных молекулярных взаимодействий (~40% — аутокринные/паракринные, ~17% — внеклеточный матрикс-рецепторы, ~13% — межклеточные контакты, ~30 % — небелковый сигнал).
3. По сравнению с версией v1, CellChatDB v2 добавляет более 1000 типов информации о взаимодействии белков и небелков, таких как метаболизм и синаптическая передача сигналов.
4. Для ключевых молекул, которые не оказывают прямого влияния на данные, этот инструмент также может предсказать экспрессию рецепторов-лигандов, а затем предсказать потенциальную связь, анализируя ключевые медиаторы/ферменты этих молекул (это для ~30% не- сигнальные взаимодействия белков) объяснение источников информации).
5. В этой версии также добавлено больше дополнительной аннотационной информации: например, ключевые слова UniProtKB (включая биологические процессы, молекулярные функции, функциональные категории, заболевания и т. д.), субклеточные местоположения и корреляции с нейротрансмиттерами.
6. Библиотеки рецепторов-лигандов человека и мышей различаются, и их необходимо устанавливать самостоятельно: CellChatDB.human/CellChatDB.mouse.

Что касается процесса ввода и подготовки данных, разработчики предоставляют различные методы обработки данных, включая матрицу conut, объект seruat, объект SingleCellExperiment, объект Anndata и т. д.
Подробный процесс подготовки можно посмотреть на официальном сайте github. Ссылка прикреплена в справочных материалах. Автор использует объекты Seurat здесь.
Шаг процесса
1.Импорт
rm(list = ls())
library(CellChat)
library(Seurat)
library(qs)
library(patchwork)
library(BiocParallel)
register(MulticoreParam(workers = 4, progressbar = TRUE))
load('scRNA.Rdata')
# Проверьте это
DimPlot(scRNA,pt.size = 0.8,group.by = "celltype",label = T)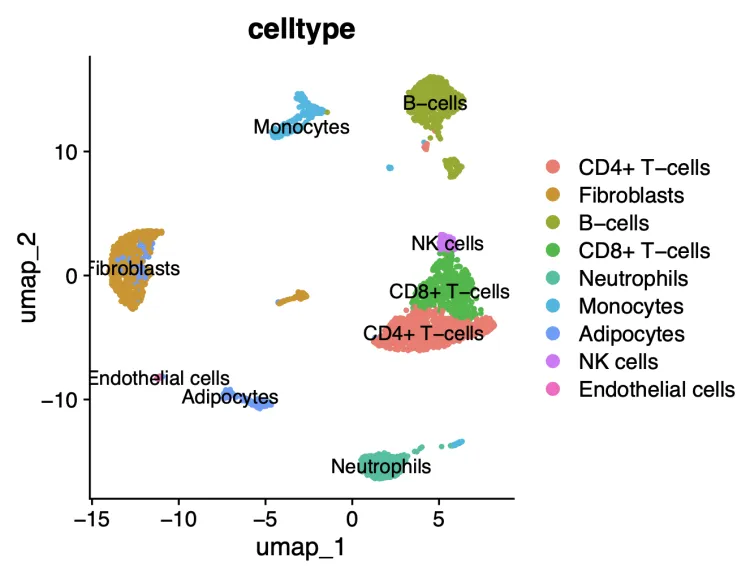
2. Предварительная обработка данных
CellChat требует ввода двух пользовательских данных: один — данные об экспрессии генов в клетке, а другой — назначенная пользователем метка ячейки.
В матрице данных экспрессии генов поведенческие гены представлены в виде ячеек.
CellChat требует нормализации данных. Если это данные подсчета, существует встроенная функция для обработки и преобразования (но обычно подсчет не используется, а используются нормализованные данные).
Перед использованием рекомендуется отсортировать данные.
data.input <- GetAssayData(scRNA, layer = 'data')
meta <- scRNA@meta.data[,c("orig.ident","celltype")]
colnames(meta) <- c("group","celltypes")
table(meta$celltypes)
meta$celltypes <- gsub(" cells|-cells", "", meta$celltypes)
table(meta$celltypes)
# Adipocytes B CD4+ T CD8+ T Endothelial Fibroblasts
# 244 667 1007 547 28 762
# Monocytes Neutrophils NK
# 311 378 87
# Рекомендуется заранее отсортировать тип ячейки~
identical(rownames(meta),colnames(data.input))
celltype_order <- c("CD4+ T","CD8+ T","B","Adipocytes","Endothelial",
"Fibroblasts","Monocytes","Neutrophils","NK")
meta$celltypes <- factor(meta$celltypes ,levels = celltype_order)
# в соответствии с meta$celltypes Сортировать по порядку
ordered_indices <- order(meta$celltypes)
# верно meta и data.input Сортировать
meta <- meta[ordered_indices, ]
data.input <- data.input[, ordered_indices]
identical(rownames(meta),colnames(data.input))3.Создайте объект Cellchat.
# построитьcellchat
cellchat <- createCellChat(object = data.input,
meta = meta,
group.by = "celltypes")
levels(cellchat@idents)
# [1] "CD4+ T" "CD8+ T" "B" "Adipocytes" "Endothelial" "Fibroblasts"
# [7] "Monocytes" "Neutrophils" "NK" 4. Создайте базу данных взаимодействия лиганд-рецептор.
CellChatDB <- CellChatDB.human # use CellChatDB.mouse if running on mouse data
showDatabaseCategory(CellChatDB)
dplyr::glimpse(CellChatDB$interaction)
# Анализ межклеточной связи с использованием CellChatDB, базы данных для конкретной среды.
# Секретный используется в примере Signaling
# CellChatDB.use <- subsetDB(CellChatDB, search = "Secreted Signaling", key = "annotation")
# Only uses the Secreted Signaling from CellChatDB v1
# CellChatDB.use <- subsetDB(CellChatDB, search = list(c("Secreted Signaling"), c("CellChatDB v1")), key = c("annotation", "version"))
# Анализ межклеточной связи с использованием всех данных CellChatDB.
CellChatDB.use <- CellChatDB
# Установите базу данных, которая будет использоваться во встроенном сотовом чате.
cellchat@DB <- CellChatDB.use5. Предварительная обработка данных экспрессии для анализа межклеточных коммуникаций.
cellchat <- subsetData(cellchat)
# future::plan("multisession", workers = 1) # do parallel
cellchat <- identifyOverExpressedGenes(cellchat)
cellchat <- identifyOverExpressedInteractions(cellchat)
# По умолчанию cellchat использует object@data.signaling для определения сети.
# Кроме того, предусмотрена функция projectData для проецирования генов в приезжатьPPI. Разработчик сказал, что PPI не вызывает или вызывает очень мало псевдокоммуникаций.
cellchat <- projectData(cellchat, PPI.human)
Как получить информацию о межклеточной связи:
Этот инструмент определяет, какие ячейки присутствуют в анализируемых данных.Сверхэкспрессированный лиганд или рецептор,а затем определить этиСверхэкспрессия лигандов или рецепторов и информация о взаимодействии соответствующих взаимодействующих молекул。Данные экспрессии генов могут быть сопоставленыприезжатьбелок-белокв интерактивной сети。
Как предсказать межклеточную связь:
1、Cellchatкаждомуситуация взаимодействия и вероятностьи провести проверку перестановок(permutation тест), чтобы сделать вывод о сотовой связи в биологическом смысле.
2. CellChat моделирует вероятность межклеточной коммуникации, используя законы масс для объединения априорно известных знаний об экспрессии генов с взаимодействиями между лигандами сигнальной трансдукции, рецепторами и их кофакторами. Среди них закон массы (закон of mass действие) означает: в контексте межклеточной коммуникации,Закон действия масс используется для описания вероятности взаимодействия лиганда и рецептора. Конкретно,Когда лиганд и один или несколько рецепторов связываются,Эффективность передачи сигнала можно считать пропорциональной произведению концентрации лиганда и рецептора. это означает,Чем выше уровень экспрессии лиганда и рецептора,Чем больше возможность межклеточной передачи сигналов。
3、Количество пар лиганд-рецептор основано на средней экспрессии генов на группу клеток.。Метод расчета по умолчанию:“trimean”,Это строгий подход。Если вы хотите быть более расслабленным, вы можете установить type="truncatedMean" и Trim=0.1 в ComputeCommunProb.,Например, ComputeAveExpr(cellchat, Features = c("CXCL12","CXCR4"), type = "truncatedMean", Trim = 0.1)。
4. Предполагая, что транскриптомы отдельных клеток анализируются без разделения клеток, CellChat также может учитывать каждую отдельную клетку в вероятностном анализе, исходя из предположения, что богатые популяции клеток имеют тенденцию коллективно посылать более сильные сигналы, чем редкие популяции клеток. Влияние доли клеток. в клеточной популяции (это может означать, что таким образом можно анализировать данные без четко определенных типов клеток)
6. Прогнозирование сетей межклеточной связи
# Ключевым параметром этого анализа является тип, то есть метод, используемый для расчета средней экспрессии гена для каждой группы клеток.
# По умолчанию введите = «triMean» производит меньшее, но более сильное взаимодействие.
# При настройке type = “truncatedMean” час,Должен ли вертрим установить значение,что приводит к большему количеству взаимодействий。
cellchat <- computeCommunProb(cellchat, type = "triMean",raw.use = TRUE)
# следующее
# computeAveExpr(cellchat, features = c("CXCL12","CXCR4"),
# type = "truncatedMean",trim = 0.1)
# min.cells — для установки порога, минимум — 10 ячеек, необходимых для вывода связи (можно настроить)
cellchat <- filterCommunication(cellchat, min.cells = 10)7. Сделайте вывод о межклеточной коммуникации на уровне сигнального пути.
# CellChat суммирует всю информацию, относящуюся к каждому сигнальному пути. Вероятность связи для расчета вероятности связи на уровне пути прохождения сигнала.
# NB:Каждый предполагаемый лиганд-рецепторверносеть межклеточной связии Каждый путь прохождения сигнала хранится отдельно в слоте.'net'и'netP'середина。
cellchat <- computeCommunProbPathway(cellchat)
# Вычислительно-агрегированные сети межклеточной связи
# Для интерактивной сетевой интеграции можно установить источник и цель. Если они не установлены, все будут установлены по умолчанию.
cellchat <- aggregateNet(cellchat)
#Извлечение данных, функция subsetCommunication
df.net <- subsetCommunication(cellchat)
qsave(cellchat,"cellchat.qs")
save(df.net,file = "df.net.Rdata")
head(df.net)Информация о взаимодействии, обнаруженная различными типами ячеек, извлекается из файла df.net.
8. Визуализируйте сети межклеточной связи.
Круговая диаграмма показывает количество взаимодействий.
groupSize <- as.numeric(table(cellchat@idents))
par(mfrow = c(1,2), xpd=TRUE)
netVisual_circle(cellchat@net$count, vertex.weight = groupSize,
weight.scale = T, label.edge= F, title.name = "Number of interactions")
netVisual_circle(cellchat@net$weight, vertex.weight = groupSize,
weight.scale = T, label.edge= F, title.name = "Interaction weights/strength")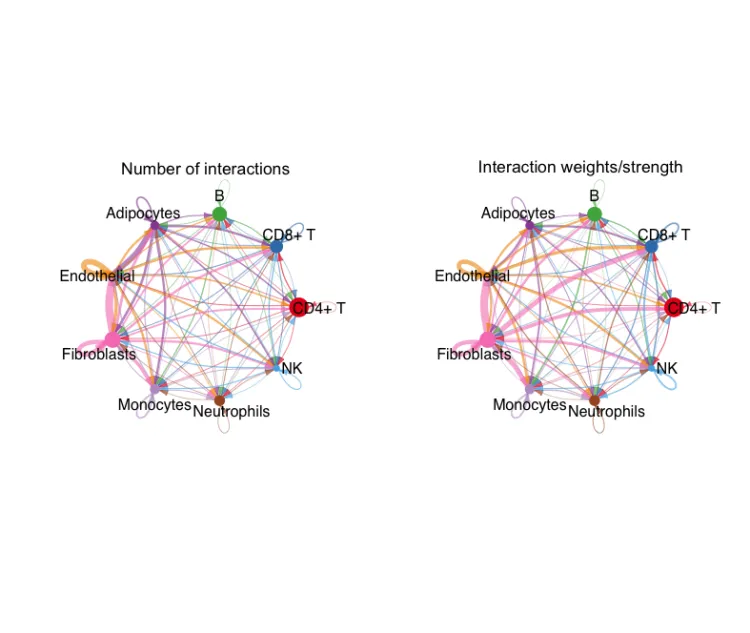
Эти два рисунка показывают количество взаимодействий и вес взаимодействий соответственно. Каждый цвет представляет отдельную ячейку,Стрелки обозначают порядок,Толщина линии представляет количество/вес.
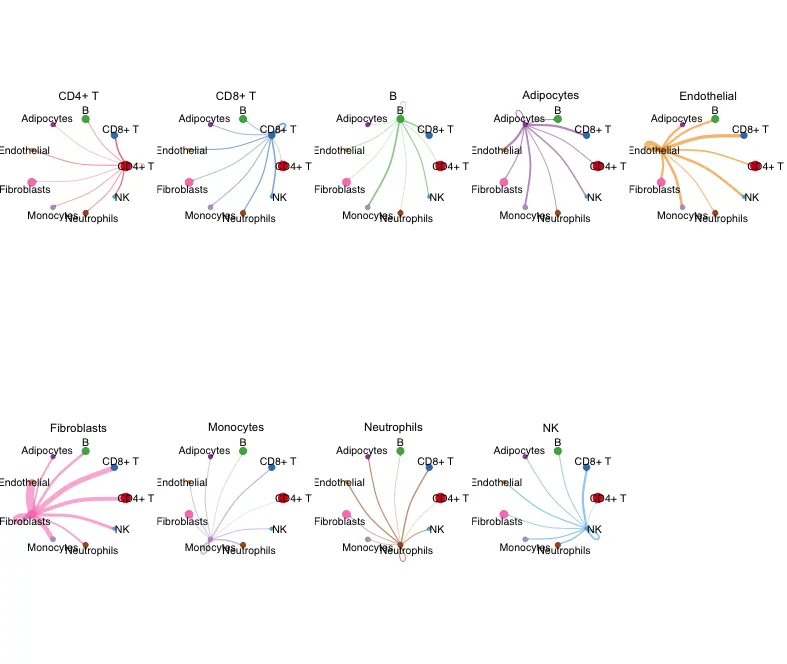
9. Круговая диаграмма разделения подгрупп
Показывает весовое взаимодействие каждого типа клеток как источника с другими типами клеток.
mat <- cellchat@net$weight
par(mfrow = c(2,5), xpd=TRUE,mar = c(1, 1, 1, 1))
for (i in 1:nrow(mat)) {
mat2 <- matrix(0, nrow = nrow(mat), ncol = ncol(mat), dimnames = dimnames(mat))
mat2[i, ] <- mat[i, ]
netVisual_circle(mat2, vertex.weight = groupSize,
weight.scale = T, arrow.size=0.05,
arrow.width=1, edge.weight.max = max(mat),
title.name = rownames(mat)[i]
)
}

В простом предложении: дальше можно делать различные визуализации.

Иерархия plot) :Фигура состоит из двух частей,В левой части показановерно Группа клеток, интересующая исследователя(vertex.receiver)аутокринного/паракринная передача сигналов,Справа показана аутокринная и паракринная передача сигналов для других вторичных клеток, представляющих интерес в наборе данных.
идиаграмма аккордов(Chord diagram) : предоставляет netVisual_chord_cell и netVisual_chord_gene Две функции. netVisual_chord_cell Для визуализации межклеточной связи между разными группами клеток etVisual_chord_gene; Используется для визуализации межклеточной коммуникации, опосредованной множеством рецепторов-лигандов или сигнальных путей.
Объяснение цвета/веса края, цвета/размера/формы узла:цвет краяи Цвет источника сигнала соответствует,Вес ребра пропорционален силе взаимодействия.
10. Иерархическая диаграмма/круговая диаграмма/диаграмма хорд/тепловая карта
cellchat@netP$pathways
levels(cellchat@idents)
vertex.receiver = seq(1:3) # a numeric vector
pathways.show <- "COLLAGEN"
# Hierarchy plot
netVisual_aggregate(cellchat, signaling = pathways.show,
vertex.receiver = vertex.receiver,layout= "hierarchy")
# vertex.size = groupSize)
# circle plot
netVisual_aggregate(cellchat, signaling = pathways.show,layout = "circle")
# Chord diagram
par(mfrow=c(1,1))
netVisual_aggregate(cellchat, signaling = pathways.show, layout = "chord")
#диаграмма сгруппированных аккордов
levels(cellchat@idents)
group.cellType <- c(rep("T/B", 3),
"Adipocytes","Endothelial","Fibroblasts",
"Monocytes","Neutrophils", "Neutrophils")
names(group.cellType) <- levels(cellchat@idents)
netVisual_chord_cell(cellchat, signaling = pathways.show,
group = group.cellType,
title.name = paste0(pathways.show, " signaling network"))
# heatmap
par(mfrow=c(1,1))
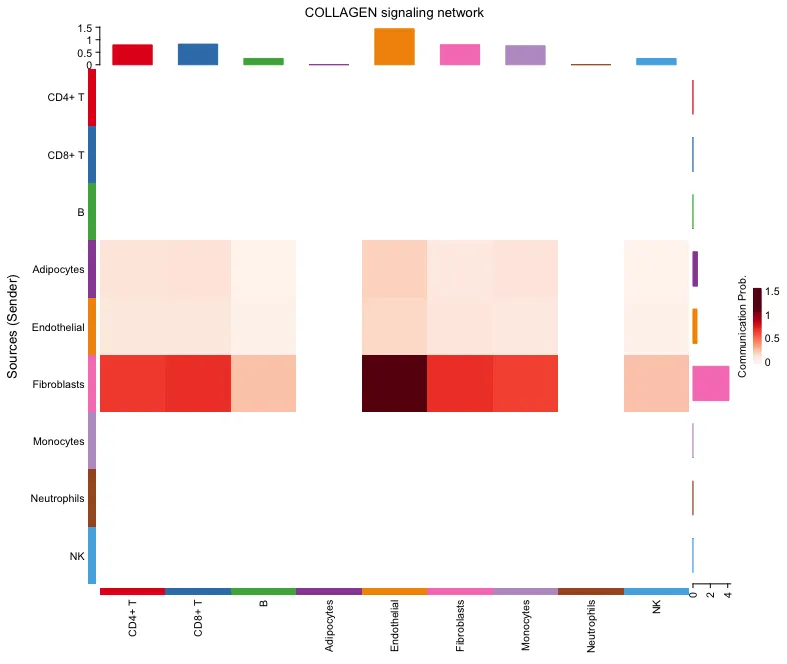
netVisual_heatmap(cellchat, signaling = pathways.show, color.heatmap = "Reds")Диаграмма иерархии
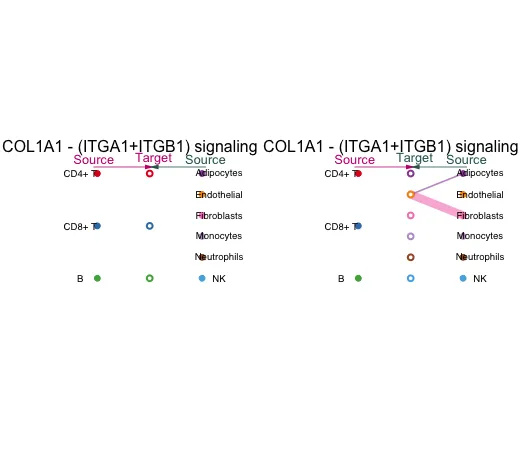
vertex.receiver устанавливает тип ячейки источника (закрашенный круг),Открытый круг представляет цель. В левой половине изображения круг, обозначающий вершину.приемник, сначала располагается посередине.,Показаны различные типы клетокверноэтотCD4+T,Роль CD8+TиB-клеток,В правой половине изображения источник клеток находится посередине.,Показаны различные типы клетокверно Что делают другие клетки,Толщина линии представляет силу взаимодействия.
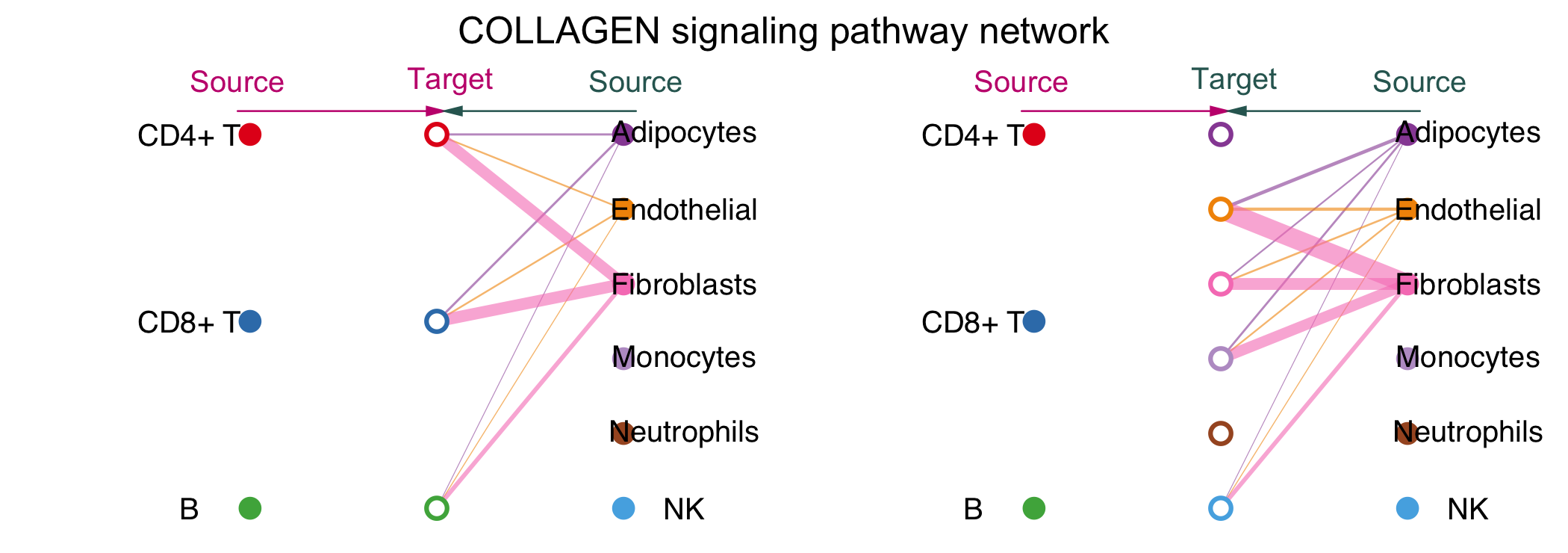
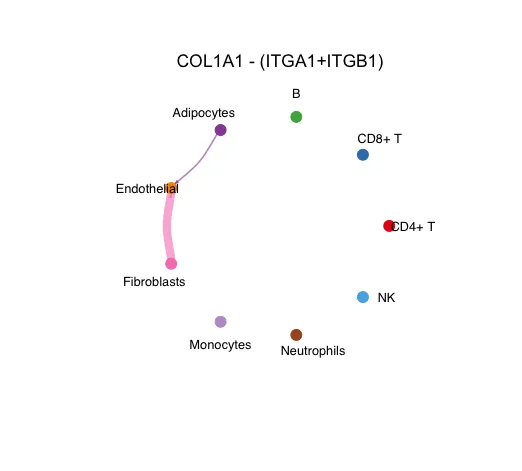
Круговая диаграмма
Разные цвета представляют разные типы клеток, комбинированные стрелки обозначают направление взаимодействия, а толщина представляет интенсивность взаимодействия.
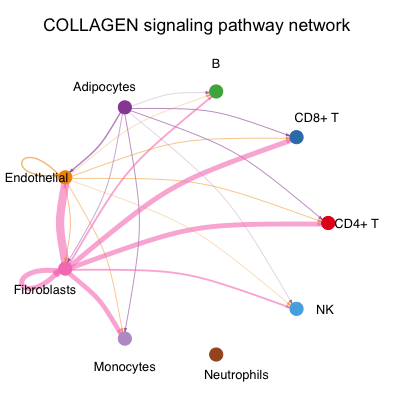
диаграмма аккордов
Интерпретация изображения аналогична изображению выше.
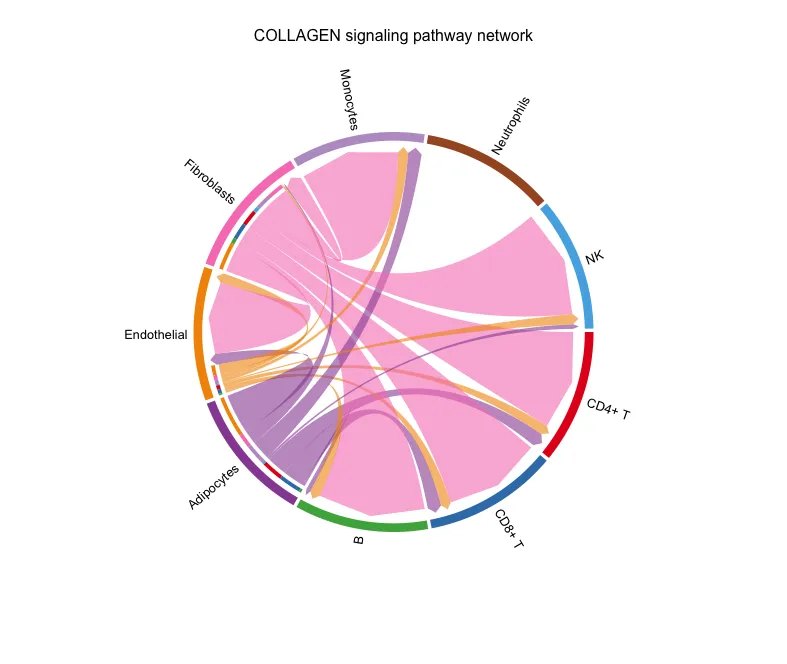
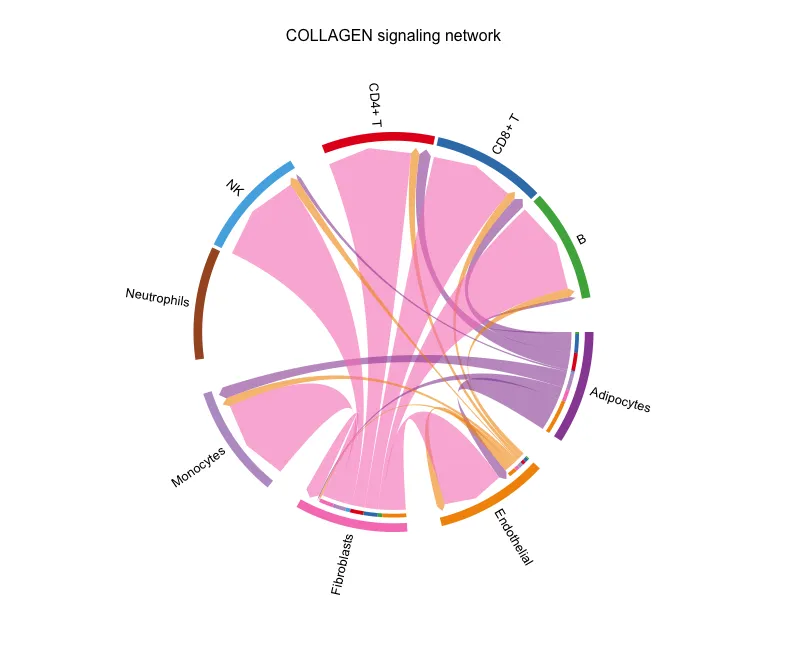
диаграмма сгруппированных аккордов
верноTиBНастроить единообразноT/B,Таким образом, ячейки, принадлежащие группе T/B на графике, приобретают одинаковый цвет (здесь розовый).
тепловая карта
Строки Строка(Источники - Отправитель):тепловая карта представляют различные типы клеток, которые служат отправителями сигналов (Источниками).
Столбцы Столбцы (Цели - Приемник):тепловая карта представляют разные типы ячеек.,этотклетки, служащие сигналамиполучатель(Target)。
Глубина цвета в Цветовые соединения:тепловая карта представляет вероятность связи. Чем темнее цвет,Указывает, что чем выше вероятность связи,Это означает, что передача сигнала между ячейкой-отправителем и ячейкой-получателем сильнее.
Проблема связи: Цветная полоса справа является эталоном для цветового сопоставления. Темно-красный цвет на рисунке указывает на более высокую вероятность связи (ближе к 1,5), а более светлые цвета указывают на меньшую вероятность связи (ближе к 0 или ниже).
Диапазон верхних значений (0–1,5): диапазон значений, показанный в верхнем красном поле, представляет собой вероятность связи, то есть вероятность связи, соответствующую различным цветам. Это значение представляет собой вероятность связи определенного сигнального пути между разными типами клеток. 0 указывает на отсутствие связи, а 1,5 указывает на относительно высокую вероятность связи.
Числовой диапазон справа (0–4): Числовой диапазон, отображаемый в красном поле справа, представляет количество путей связи (Количество коммуникаций). 0 означает отсутствие пути связи, 4 означает, что несколько путей связи рассчитываются и отображаются на тепловой карте.
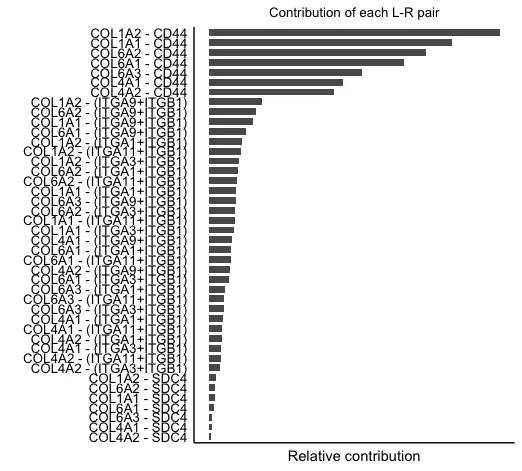
11. Рассчитайте и визуализируйте вклад рецепторов-лигандов в сигнальные пути.
# Рассчитать распределение-рецепторверно Вклад сигнальных путей и визуализация
netAnalysis_contribution(cellchat, signaling = pathways.show)
# Визуализация по одному лиганду-рецепторверноопосредованная межклеточная коммуникация
pairLR <- extractEnrichedLR(cellchat, signaling = pathways.show,
geneLR.return = FALSE)
LR.show <- pairLR[1,] # show one ligand-receptor pair
# Hierarchy plot
vertex.receiver = seq(1,3) # a numeric vector
netVisual_individual(cellchat, signaling = pathways.show,
pairLR.use = LR.show,
vertex.receiver = vertex.receiver,
layout = "hierarchy")Судя по этим данным, это включает вклад лиганд-рецепторов в путь коллагена.
Визуализируйте одно из условий передачи сигналов лиганд-рецептор.
Диаграмма иерархии:
круговая диаграмма
12.Официальный код хранения изображений.
Если честно, пользоваться картой непросто. Лучше настроить карту самостоятельно.
# Access all the signaling pathways showing significant communications
pathways.show.all <- cellchat@netP$pathways
# check the order of cell identity to set suitable vertex.receiver
levels(cellchat@idents)
vertex.receiver = seq(1,3)
for (i in 1:length(pathways.show.all)) {
# Visualize communication network associated with both signaling pathway and individual L-R pairs
netVisual(cellchat, signaling = pathways.show.all[i], vertex.receiver = vertex.receiver, layout = "hierarchy")
# Compute and visualize the contribution of each ligand-receptor pair to the overall signaling pathway
gg <- netAnalysis_contribution(cellchat, signaling = pathways.show.all[i])
ggsave(filename=paste0(pathways.show.all[i], "_L-R_contribution.pdf"), plot=gg, width = 3, height = 2, units = 'in', dpi = 300)
}
Систематический анализ сетей межклеточной связи:
Чтобы лучше понять сложные сети межклеточной связи, CellChat Для количественного измерения и анализа этих сетей использовался ряд математических и вычислительных методов из разных областей. К этим методам относится теория графов. теория), распознавание образов (паттерн признание) и многообразное обучение (многообразное learning)。
Определения трех вышеуказанных методов:
● Теория графов: используется для построения и анализа структурных свойств сетей межклеточной связи, таких как связность, центральность и т. д.
● Распознавание образов: используется для автоматической идентификации и классификации конкретных шаблонов общения на основе данных связи.,Помогите обнаружить конкретные лиганды-рецепторвернофункциональная роль。
● Многообразное обучение: используется для уменьшения размерности и визуализации многомерных коммуникационных данных, помогая выявить сложные нелинейные коммуникационные отношения.
- CellChat может использовать индикаторы центральности в сетевом анализе, чтобы определить, какие ячейки являются основными источниками сигналов, целями, посредниками и факторами влияния в конкретной сигнальной сети.
- Это можно сделать с помощью метода распознавания образов.,Прогнозирование критических входных и выходных сигналов для определенных типов клеток,и скоординированные ответы между различными типами клеток.
- CellChat также может определять меры сходства.,И выполнять многообразное обучение с точки зрения функций и топологии.,Групповые сигнальные пути.
- Это также может пройтиверно Совместное многообразное обучение с несколькими сетями,Различать консервативные (общие) и контекстно-зависимые сигнальные пути.
СуммируяCellChat может рассчитывать несколько показателей центральности сети для каждой популяции ячеек.,признанныйв клеткахсеть связисерединаглавный отправитель、получатель、медиатор и влиятельный человек。Конкретно,Разработчики использовали несколько метрик из взвешенных направленных сетей.,В том числе вне степени, в степень, поток между (поток посредничество) и информационная центральность (информационная centrality),чтобы определить, какие популяции клеток являются первичными отправителями сигнала соответственно、получатель、Медиатор и влиятельный человек.
13. Дальнейшая визуализация результатов на основе лиганд-рецептора
Пузырьковый графикnetVisual_bubble показывает все важные взаимодействия между некоторыми группами объектов и другими группами объектов.
# Необходимо указать источник и цель
# source.use — это линия ячеек, которая отправляет сигнал, а target.use — это линия ячеек, которая получает сигнал.
levels(cellchat@idents)
netVisual_bubble(cellchat, sources.use = seq(1:3),
targets.use = c(4:10), remove.isolate = FALSE)
ggsave("bubbleplot.pdf",width = 8,height = 16)
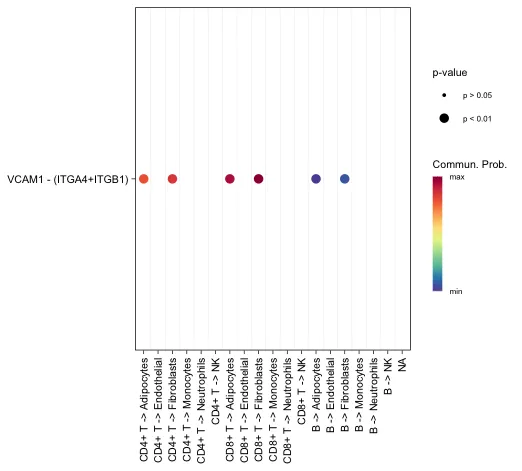
# Вы также можете добавить параметры сигнализации для отображения конкретных лигандов.
cellchat@netP$pathways
netVisual_bubble(cellchat, sources.use = seq(1:3),
targets.use = c(4:10),
signaling = c("VCAM"),
remove.isolate = FALSE)
# Индивидуализированный дисплей входных сигналов
# pairLR.use <- extractEnrichedLR(cellchat, signaling = c("CCL","CXCL","FGF"))
# netVisual_bubble(cellchat, sources.use = c(1:3),
# targets.use = c(4:10),
# pairLR.use = pairLR.use,
# remove.isolate = TRUE)
# Вы можете установить порядок по оси X, добавив следующие параметры
# sort.by.target = T
# sort.by.source = T
# sort.by.source = T, sort.by.target = T
# sort.by.source = T, sort.by.target = T, sort.by.source.priority = FALSEГоризонтальная ось представляет направление взаимодействия между различными ячейками. Вертикальная ось представляет конкретные пары лиганд-рецептор. Разные цвета точек обозначают вероятности связи. Размер точек представляет собой значение P значимости общения.
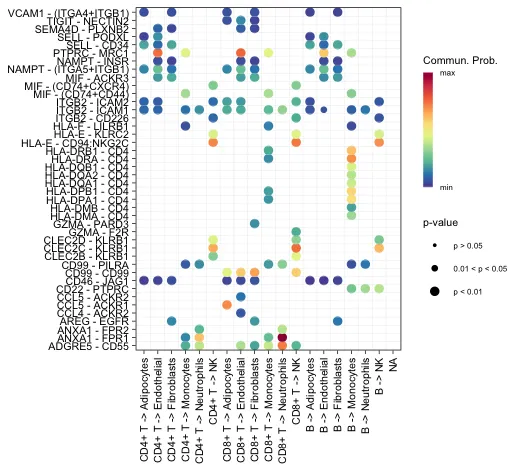
Демонстрация состояния конкретного пути
14. Chord diagram
# При рисовании здесь рекомендуется установить одиночное выделение или уменьшить количество взаимодействующих ячеек.
# Большая часть контента приведет к отсутствию изображений.
cellchat@netP$pathways
netVisual_chord_gene(cellchat, sources.use = c(1:3),
targets.use = c(4:10),
#signaling = c("COLLAGEN"),
lab.cex = 0.5,
legend.pos.y = 30)
# Отображает все важные сигнальные пути от определенных групп клеток (sources.use) к другим группам клеток (targets.use).
netVisual_chord_gene(cellchat, sources.use = c(1:3),
targets.use = c(4:10),
slot.name = "netP",
legend.pos.x = 10)
# NB: Please ignore the note when generating the plot such as “Note: The first link end is drawn out of sector ‘MIF’.”. If the gene names are overlapped, you can adjust the argument small.gap by decreasing the value.Когда пользователи выполняют собственный анализ, не отображайте слишком много ситуаций лиганд-рецептор. Слишком большое количество приведет к ситуации, показанной на рисунке.
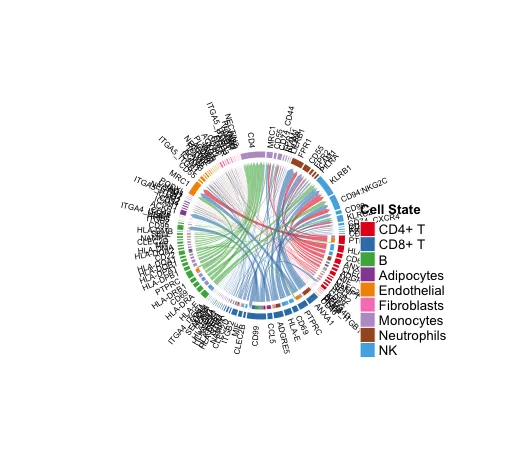
Показывает все важные сигнальные пути от определенных групп клеток (sources.use) к другим группам клеток (targets.use).
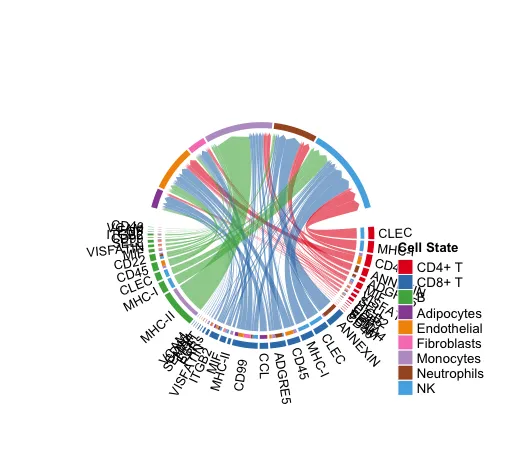
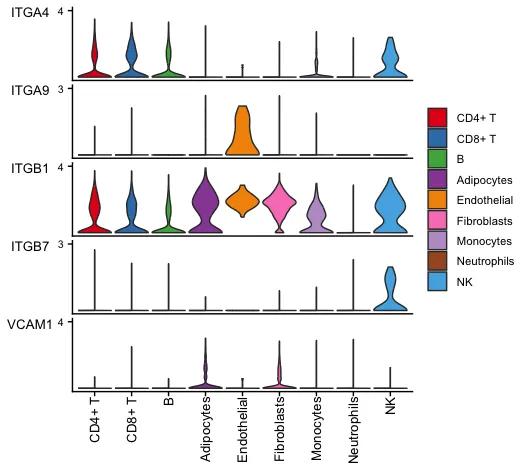
15. Используйте скрипку/точечный график, чтобы построить график экспрессии генов сигнальных путей.
# CellChat можно построить с помощью функции-оболочки SeuratplotGeneExpression и L-R верноили фаза пути сигналазакрывать Распределение экспрессии генов генов сигнальной трансдукции。
# Эта функция обеспечивает “violin”、“dot”、“bar” Три типа визуализации.
# Или пользователь может использовать extractEnrichedLR извлечено и выведено L-R верноили фаза пути сигналазакрыватьгены сигнальной трансдукции,Затем экспрессию генов строят с использованием Seurat или других программных пакетов.
plotGeneExpression(cellchat, signaling = "VCAM",
обогащенный.only = ИСТИНА,
тип = "скрипка")Отобразить экспрессию ключевых генов определенного пути в разных клеточных линиях.
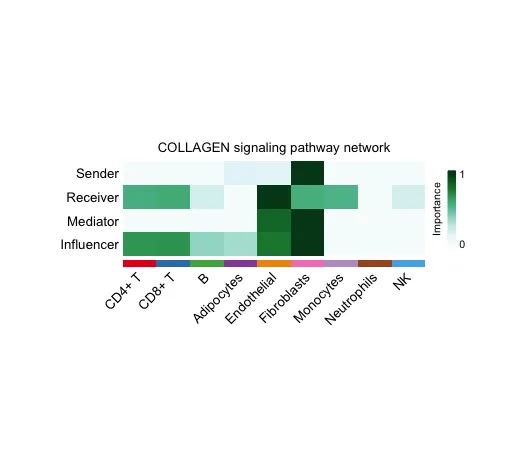
16. Систематический анализ сетей межклеточной связи для расчета и визуализации показателей центральности сети.
# Compute the network centrality scores
cellchat <- netAnalysis_computeCentrality(cellchat,
slot.name = "netP")
netAnalysis_signalingRole_network(cellchat, signaling = pathways.show,
width = 8, height = 2.5, font.size = 10)Строки (Отправитель, Получатель, Медиатор, Влиятель): Строки представляют роли, которые играют различные типы клеток в сети сигнальных путей:
● Отправитель: отправитель сигнала, то есть типы ячеек, которые являются основными отправителями сигнала.
● Приемник: получатель сигнала, то есть типы ячеек, которые являются основными приемниками сигнала.
● Медиатор: Медиатор, то есть тип клеток, который играет роль посредника в процессе передачи сигнала.
● Влиятель: Влиятель, то есть тип клеток, который оказывает существенное влияние на процесс коммуникации во всей сети сигнальных путей.
Столбцы (различные типы клеток): Столбцы представляют определенные типы клеток, такие как CD4+ Т-клетки, В-клетки, фибробласты и т. д.
Интенсивность цвета (важность). Интенсивность цвета указывает на важность каждого типа клеток в определенной роли. Чем темнее цвет, тем важнее в этой роли тип клетки (например, чем больше интенсивность или частота передачи сигнала, тем менее важен в этой роли тип клетки);
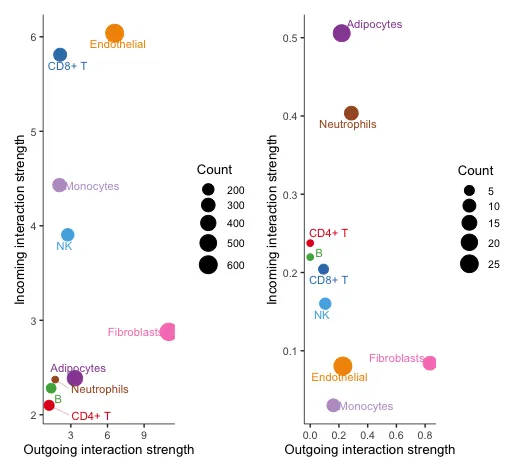
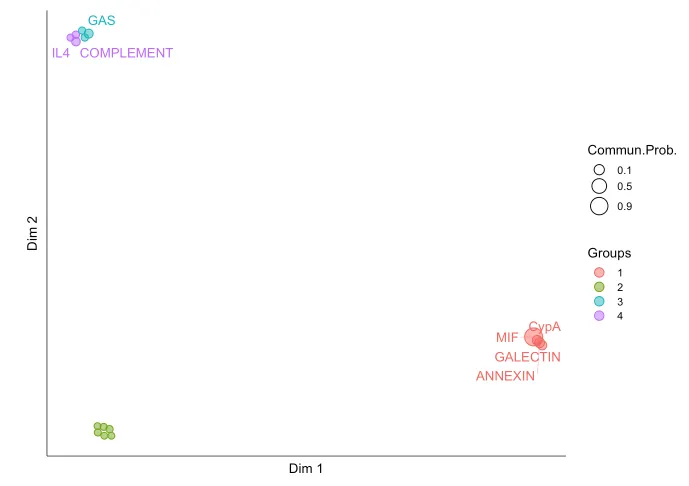
17 Визуализируйте основных отправителей (Источник) и получателей (цели) в двухмерном пространстве.
# Анализ сигнальных ролей в агрегированных сетях межклеточной коммуникации всех сигнальных путей.
p1 <- netAnalysis_signalingRole_scatter(cellchat)
# верно Анализ сигнальных эффектов в конкретных сетях межклеточной связи
p2 <- netAnalysis_signalingRole_scatter(cellchat, signaling = c("CXCL", "CCL"))
p1 + p2Левая часть представляет собой комплексную оценку всех сигналов. Видно, что фибробласты очень сильны в выдаче сигналов, а эндотелиальные очень сильны в приеме сигналов.
Правая часть представляет оценку конкретного сигнала.,Здесь был выбран сигнал рецептора лиганда CXCLиCCL. Вы можете увидеть, являются ли приезжающие или фибробласты очень сильными с точки зрения выходного сигнала.,Адипоциты очень хорошо воспринимают сигналы.
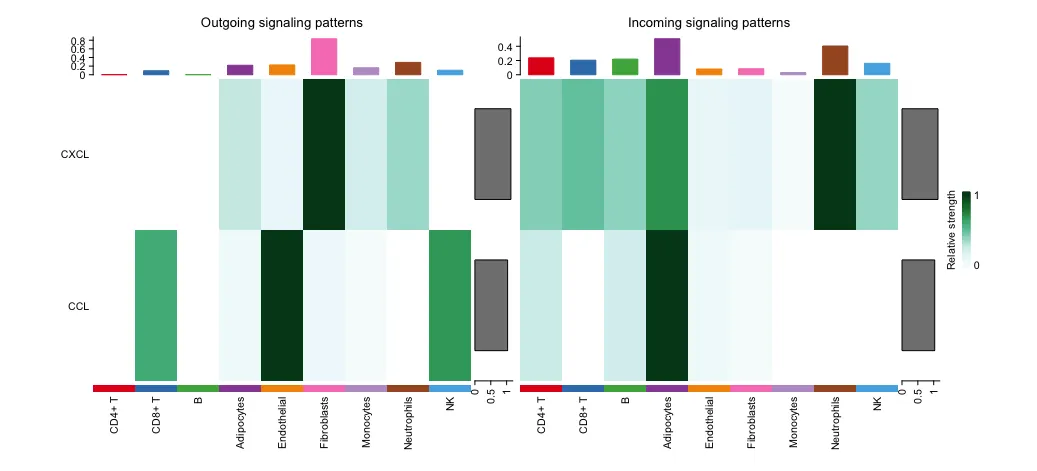
18. Определите сигналы, которые больше всего способствуют выходным или входным сигналам определенных популяций клеток.
# pdf("ht1.pdf",width = 12,height = 18)
# ht1 <- netAnalysis_signalingRole_heatmap(cellchat, pattern = "outgoing")
# dev.off()
#
# pdf("ht2.pdf",width = 12,height = 18)
# ht2 <- netAnalysis_signalingRole_heatmap(cellchat, pattern = "incoming")
# dev.off()
# ht1 + ht2
# class(ht1)
# специфическая сигнализация
htout <- netAnalysis_signalingRole_heatmap(cellchat,
pattern = "outgoing",
signaling = c("CXCL", "CCL"))
htout
htcome <- netAnalysis_signalingRole_heatmap(cellchat,
pattern = "incoming",
signaling = c("CXCL", "CCL"))
htcomeНе забывайте фильтровать, не используйте их все, картинка будет некрасивой.

Помимо изучения подробной связи отдельных сигнальных путей, также возможно дальнейшее изучение того, как множественные популяции клеток и сигнальные пути действуют согласованно.
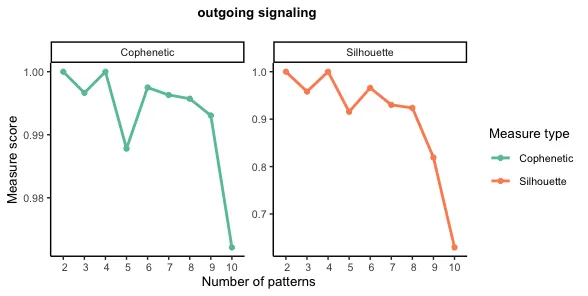
CellChat использует подход распознавания образов для выявления этих глобальных коммуникационных шаблонов. По мере увеличения количества паттернов могут появиться некоторые избыточные паттерны, что может затруднить интерпретацию коммуникативных паттернов. Поэтому мы выбрали для анализа пять режимов по умолчанию. Вообще говоря, когда количество паттернов превышает 2, это более биологически значимо.
Кроме того, разработчик предоставляет selectK Функция угадывания соответствующего количества шаблонов. Эта функция основана на двух существующих NMF R Индикаторы, реализованные в пакете: Кофенетик и Silhouette。этот Два показателя пройденыверно Иерархическая кластеризация матрицы консенсуса,Измеряет стабильность определенного количества шаблонов。В серии объемов шаблона, когда Cophenetic(Cophenetic коэффициент корреляции) и Silhouette Когда значение (коэффициента силуэта) начинает внезапно падать, это обычно указывает на количество подходящих режимов.(Cophenetic Чем ближе значение к 1. Указывает, что дерево кластеризации может лучше поддерживать соотношение расстояний между исходными точками данных и тем лучше результат кластеризации. если Cophenetic Низкое значение указывает на то, что дерево кластеризации может быть искажено и не может хорошо отражать исходную структуру данных; Silhouette Значение является мерой того, насколько разумной является отдельная точка данных в кластере. Он оценивает расстояние между точкой данных и точкой кластера, в котором она расположена, по сравнению с расстоянием от ближайших других кластеров. Silhouette Чем ближе значение к 1, тем лучше это указывает на то, что точка данных соответствует кластеру, в котором она расположена, и значительно отличается от других ближайших кластеров).

Паттерны отправки показывают, как сигнальные клетки — клетки, из которых они происходят, — координируются друг с другом и как они работают с определенными сигнальными путями, чтобы управлять коммуникацией. Режим приема показывает, как клетки-мишени (т. е. клетки, принимающие сигнал) координируют друг друга и как они реагируют на внешние сигналы во взаимодействии с определенными сигнальными путями.
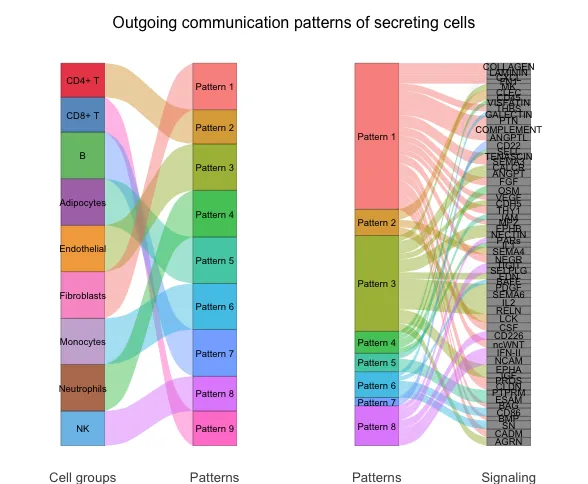
Для режима отправки (или приема) матрица шаблонов популяции ячеек в матрице вероятности передачи (или приема) межсотовой связи, полученной путем матричного разложения. W представляет, как эти группы клеток координально отправляют (или получают) сигналы, а также матрицу шаблонов сигнальных путей. H Представляет, как эти сигнальные пути работают вместе для отправки (или получения) сигналов. Чтобы визуализировать, как скрытые закономерности связаны с популяциями клеток и парами лиганд-рецептор или сигнальными путями, разработчики использовали речные графики. Сначала поставь W каждая строка H Каждый столбец нормирован к [0,1] в пределах диапазона, а затем W и H Элементы, размер которых меньше порогового значения (по умолчанию 0,5), устанавливаются в ноль. Такое определение порога может выявить наиболее обогащенные популяции клеток и сигнальные пути, связанные с каждым предполагаемым паттерном. пороговая матрица W и H Используется в качестве входных данных для создания графика реки.
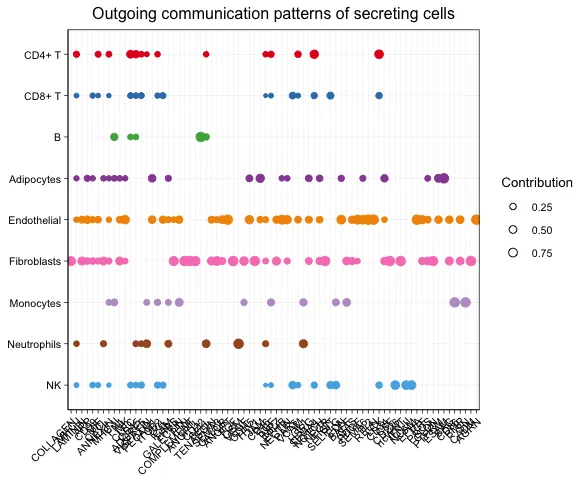
Кроме того, чтобы напрямую коррелировать клеточные популяции с их обогащенными сигнальными путями, разработчики будут W и H Элементы, размер которых меньше порога, устанавливаются в ноль (порог по умолчанию: 1/R, где R — количество потенциальных шаблонов). Используя менее строгие пороговые значения, можно получить более обогащенные сигнальные пути, связанные с каждой популяцией клеток. добавив W и H Умножьте каждую популяцию клетокверно Оценка вклада каждого сигнального пути,Разработчик строит точечный график,Размер точки и доля вклада пропорциональны,Показать связь между клеточными популяциями и их обогащенными сигнальными путями. Пользователи также могут снизить пороговые значения параметров, чтобы выявить более сложные сигнальные пути, связанные с каждой популяцией клеток.
авторверноэтот Понимание содержимого блокаПомимо изучения взаимодействия лиганд-рецептор/пути между различными клеточными линиями, разработчики,Команда также надеется соединить различные клетки с помощью общей информации о взаимодействии лиганд-рецептор/пути (расширяющейся от «частичной» до «целой»).,Поэтому неотрицательное матричное разложение используется для определения того, сколько «важных» признаков включено в целое.,И соединить эти характеристики с условиями отправки/приема сигнала разных линий сот.。
19. Определить общие закономерности коммуникации/изучить, как несколько типов клеток и сигнальных путей работают вместе.
outgoing
library(NMF)
library(ggalluvial)
selectK(cellchat, pattern = "outgoing")
# Когда количество режимов вывода равно 9, значение Cophenetic и значение Silhouette начинают внезапно уменьшаться.
nPatterns = 9
cellchat <- identifyCommunicationPatterns(cellchat,
pattern = "outgoing",
k = nPatterns)
# river plot
netAnalysis_river(cellchat, pattern = "outgoing")
# dot plot
netAnalysis_dot(cellchat, pattern = "outgoing")анализ значения k
Режим ячейки после установки определенного значения K (Cell Patterns)и Режим связи Patterns) результаты корреляционного анализа.
Левое изображение — режим ячейки (ячейка Patterns) :
Дендрограмма: показывает сходство или взаимосвязь между различными типами клеток. Более близкие разветвления указывают на то, что эти типы клеток более похожи по своим моделям общения.
Цветовой блок (Тепловая карта): указывает на то, что каждый тип ячеек имеет разные шаблоны. Вклад или участие в (Шаблон 1 приезжать Образец 9). Чем темнее цвет (например, красный), тем больше вклад этого типа клеток в этот узор.
Правое изображение – Режим связи (Связь Patterns) :
Дендрограмма: показывает сходство между различными способами общения.
Цветовой блок (тепловая карта): представляет вклад различных сигнальных путей (таких как GAL, COMP, SPP1 и т. д.) в разных режимах. Чем темнее цвет (например, красный), тем больший вклад этого сигнального пути в этот паттерн.
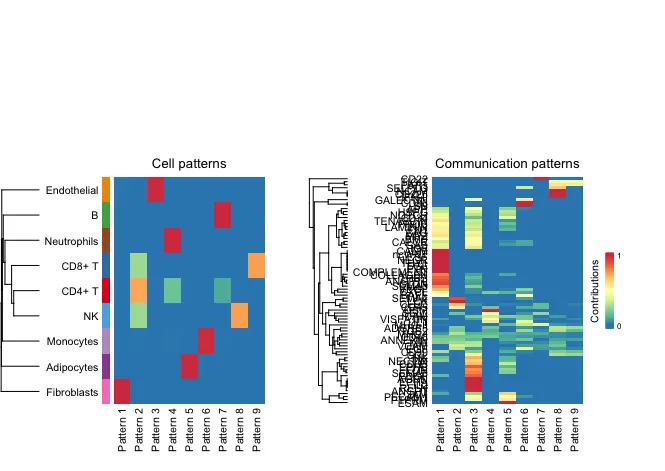
Схема реки
Точечная диаграмма
incoming
selectK(cellchat, pattern = "incoming")
# Когда количество режимов вывода равно 6, значение Cophenetic и значение Silhouette начинают внезапно падать.
nPatterns = 8
cellchat <- identifyCommunicationPatterns(cellchat,
pattern = "incoming",
k = nPatterns)
# river plot
netAnalysis_river(cellchat, pattern = "incoming")
# dot plot
netAnalysis_dot(cellchat, pattern = "incoming")Изображение здесь не будет отображаться.

CellChat способен количественно оценить сходство между всеми важными сигнальными путями и сгруппировать их по сходству в сетях сотовой связи. Группировка отображается на основе функционального/структурного сходства.
В основном анализ функционального сходствазакрывать Примечаниеотправитель сигналаиполучательсходство между。Высокое функциональное сходство означает, что основныеотправитель сигналаиполучательсхожи в разных сигнальных путях。этотобычно указываетэтот Некоторые сигнальные пути могут иметь схожие биологические функции.,Или их роль избыточна.
Анализ структурного сходства в основном касается общей структуры сигнальной сети Примечание.,Независимо от конкретного сходства между отправителем и получателем. Этот аналитический подход фокусируется на топологии сигнальной сети.,Группировка осуществляется путем сравнения сходства самой сетевой структуры.
20. Многообразное обучение и классификационный анализ сигнальных сетей.
cellchat <- computeNetSimilarity(cellchat, type = "functional")
cellchat <- netEmbedding(cellchat, type = "functional")
# Запустить кластеризацию
cellchat <- runnetClustering(cellchat,
type = c("functional"),
nCores = 1)
# Visualization in 2D-space
netVisual_embedding(cellchat,
type = c("functional"),
label.size = 3.5)
# Определите группы сигналов на основе структурного сходства
cellchat <- computeNetSimilarity(cellchat, type = "structural")
cellchat <- netEmbedding(cellchat, type = "structural")
cellchat <- runnetClustering(cellchat, type = "structural",nCores = 1)
# Visualization in 2D-space
netVisual_embedding(cellchat,
type = "structural",
label.size = 3.5)
netVisual_embeddingZoomIn(cellchat,
type = c("structural"),
nCol = 2)Пути со схожими функциями/структурами будут выделены вместе~
debug:

Если вам нужны такие отчеты об ошибках, вы можете использовать два метода. Один из них — запустить «cellchat». <- updateCellChat(cellchat)», второй — понизить версию igraph до версии 1.3.5.
Ссылки:
1、Identify signaling groups based on structure similarityInference and analysis of cell-cell communication using CellChat . Nat Commun. 2021 Feb 17;12(1):1088.
2、cellchat V2 :
https://github.com/jinworks/CellChat?tab=readme-ov-file
3. Одноклеточный мир:
https://mp.weixin.qq.com/s/Pz_pEv4RooGKKzNBphWnRw
4. Группа новичков Шэнсинь:
https://mp.weixin.qq.com/s/g4j7u0gHmpJH9ZA1Jp9hCw
https://mp.weixin.qq.com/s/npOyrOufgrZtivVFcvtlkQ
5. Дерево навыков создания:
https://mp.weixin.qq.com/s/9Q4rxGKFo_jD8vBkuOzTbQ
6、YuLabSMU:
https://mp.weixin.qq.com/s/wQJzTxf-hKUB_jIqLGSeZg
7. Эссе жизненного письма:
https://mp.weixin.qq.com/s/Tw6FH2vqLghfIQ7kxUthWw
Примечание:нравитьсяверно Друзья, которые сомневаются в содержании или нашли явные ошибки.,Пожалуйста, свяжитесь с серверной частью (добро пожаловать для общения).
- END -
Учебное пособие по Jetpack Compose для начинающих, базовые элементы управления и макет

Код js веб-страницы, фон частицы, код спецэффектов

【новый! Суперподробное】Полное руководство по свойствам компонентов Figma.
🎉Обязательно к прочтению новичкам: полное руководство по написанию мини-программ WeChat с использованием программного обеспечения Cursor.
[Забавный проект Docker] VoceChat — еще одно приложение для мгновенного чата (IM)! Может быть встроен в любую веб-страницу!

Как реализовать переход по странице в HTML (html переходит на указанную страницу)

Как решить проблему зависания и низкой скорости при установке зависимостей с помощью npm. Существуют ли доступные источники npm, которые могут решить эту проблему?

Серия From Zero to Fun: Uni-App WeChat Payment Practice WeChat авторизует вход в систему и украшает страницу заказа, создает интерфейс заказа и инициирует запрос заказа

Серия uni-app: uni.navigateЧтобы передать скачок значения

Апплет WeChat настраивает верхнюю панель навигации и адаптируется к различным моделям.

JS-время конвертации

Обеспечьте бесперебойную работу ChromeDriver 125: советы по решению проблемы chromedriver.exe не найдены

Поле комментария, щелчок мышью, специальные эффекты, js-код
Объект массива перемещения объекта JS

Как открыть разрешение на позиционирование апплета WeChat_Как использовать WeChat для определения местонахождения друзей
Я даю вам два набора из 18 простых в использовании фонов холста Power BI, так что вам больше не придется возиться с цветами!

Получить текущее время в js_Как динамически отображать дату и время в js

Вам необходимо изучить сочетания клавиш vsCode для форматирования и организации кода, чтобы вам больше не приходилось настраивать формат вручную.

У ChatGPT большое обновление. Всего за 45 минут пресс-конференция показывает, что OpenAI сделал еще один шаг вперед.
Copilot облачной разработки — упрощение разработки

Микросборка xChatGPT с низким кодом, создание апплета чат-бота с искусственным интеллектом за пять шагов

CUDA Out of Memory: идеальное решение проблемы нехватки памяти CUDA
Анализ кластеризации отдельных ячеек, который должен освоить каждый&MarkerгенетическийВизуализация

vLLM: мощный инструмент для ускорения вывода ИИ

CodeGeeX: мощный инструмент генерации кода искусственного интеллекта, который можно использовать бесплатно в дополнение к второму пилоту.

Машинное обучение Реальный бой LightGBM + настройка параметров случайного поиска: точность 96,67%
Бесшовная интеграция, мгновенный интеллект [1]: платформа больших моделей Dify-LLM, интеграция без кодирования и встраивание в сторонние системы, более 42 тысяч звезд, чтобы стать свидетелями эксклюзивных интеллектуальных решений.
LM Studio для создания локальных больших моделей
Как определить количество слоев и нейронов скрытых слоев нейронной сети?


