Интеграция нескольких образцов одной клетки scVI и scANVI
Я также использовал Python для создания BBKNN и Harmony:
scVI (вариационный вывод одной ячейки) — это алгоритм интеграции, основанный на глубоком обучении, который неоднократно оценивался как один из лучших алгоритмов интеграции. scVI был опубликован в журнале Nature Methods в 2018 году. Он неоднократно обновлялся и улучшался. Название статьи — «Глубокое генеративное моделирование для транскриптомики одиночных клеток».
Алгоритм scANVI (одноклеточная ANnotation с использованием вариационного вывода) аналогичен scVI. Это алгоритм, созданный той же командой. Он был опубликован в Mol Syst Biol в 2021 году. Название статьи: «Вероятностная гармонизация и аннотация одиночных данных». Данные транскриптомики клеток с глубокими генеративными моделями».
Уведомление,scVI и scANVI являются моделью глубокой генерации для анализа транскриптома одиночных клеток.,Все они являются частью пакета scvi-tools. scVI можно использовать для таких задач, как интеграция данных, уменьшение размерности, кластеризация и дифференциальное выражение.,scANVI можно использовать для полуконтролируемого аннотирования типов ячеек, то есть с использованием частично известных меток типов ячеек для определения статуса других ячеек (каждый пакет данных одной ячейки должен быть предварительно аннотирован заранее)。scANVIможно увидеть как вscVIиз База Классификатор добавлен в,Таким образом, его можно инициализировать из предварительно обученной scVIМодель.
- Учебное пособие на официальном сайте находится по адресу https://docs.scvi-tools.org/en/stable/tutorials/notebooks/harmonization.html.
- github находится по адресу https://github.com/scverse/scvi-tools.
Следует подчеркнуть, что использование scVI/scANVI для комплексного анализа более ресурсозатратно. При наличии графического процессора скорость будет в 5-10 раз выше. Если графического процессора нет, нужно быть осторожным при запуске только scVI. на процессоре. Потому что скорость работы scVI в режиме ЦП очень низкая, особенно при работе с большими выборками данных из одной ячейки.
1. Развертывание среды
mamba create -n scvi python=3.9
which python
#/home/data/fuli09/miniconda3/envs/scvi2/bin/python
/home/data/fuli09/miniconda3/envs/scvi2/bin/python -m pip install scvi-tools anndata numpy scanpy scib certifi scib-metrics pymde scvi-colab
#Please be sure to install a version of PyTorch that is compatible with your GPU (if applicable).
Также необходимо запустить в Python:
from scvi_colab import install
install()
Если необходимо подключениеJupyter notebookиспользоватьизразговаривать,нужно быть внутриscviУстановите несколько плагинов в этой среде.,Тогда ты сможешьJupyter notebookВыберите здесьscviэта среда(ps:Я используюиздаJupyterLab,иJupyter notebookда差不多из):
mamba install -y nb_conda_kernels ipykernel
python -m ipykernel install --user --name scvi --display-name scvi
JupyterLab
2. Запустите образец данных
import scanpy as sc
import scvi
from rich import print
from scib_metrics.benchmark import Benchmarker
from scvi.model.utils import mde
sc.set_figure_params(figsize=(4, 4))
%config InlineBackend.print_figure_kwargs={'facecolor' : "w"}
%config InlineBackend.figure_format='retina'
Шаг 1. Считайте данные.
adata = sc.read(
"data/lung_atlas.h5ad",
backup_url="https://figshare.com/ndownloader/files/24539942",
)
adata
AnnData object with n_obs × n_vars = 32472 × 15148
obs: 'dataset', 'location', 'nGene', 'nUMI', 'patientGroup', 'percent.mito', 'protocol', 'sanger_type', 'size_factors', 'sampling_method', 'batch', 'cell_type', 'donor'
layers: 'counts'
Шаг 2. Предварительная обработка набора данных.
Этот набор данных был обработан, как описано в рукописи scIB. Обычно модели в инструментах scvi ожидают стандартного анализа так же, как Scanpy/Seurat.
ценность Уведомлениеизда高度可变из Проблемы отбора генов。ХотяscVIиscANVIВсе гены могут использоваться во время выполнения,Но авторы обычно рекомендуют фильтровать гены для оптимальной эффективности интеграции. В то же время это также помогает устранить различия в зависимости от партии из-за экспрессии генов в конкретной партии.
В общем, пользователи могут выполнять гипервариабельный отбор генов, используя стандартный конвейер Scanpy.
adata.raw = adata # keep full dimension safe
sc.pp.highly_variable_genes(
adata,
flavor="seurat_v3",
n_top_genes=2000,
layer="counts",
batch_key="batch",
subset=True,
)
Шаг 3. Используйте scVI для комплексного анализа.
Первым шагом является создание модели на основе данных подсчета и информации о партии:
scvi.model.SCVI.setup_anndata(adata, layer="counts", batch_key="batch")
vae = scvi.model.SCVI(adata, n_layers=2, n_latent=30, gene_likelihood="nb")
#Now we train scVI. This should take a couple of minutes on a Colab session
vae.train()
Добавить результаты вobsm,названныйX_scVI:
adata.obsm["X_scVI"] = vae.get_latent_representation()
Наконец, набор данных кластеризуется и визуализируется:
sc.pp.neighbors(adata, use_rep="X_scVI")
sc.tl.leiden(adata)
adata.obsm["X_mde"] = mde(adata.obsm["X_scVI"])
sc.pl.embedding(
adata,
basis="X_mde",
color=["batch", "leiden"],
frameon=False,
ncols=1,
)
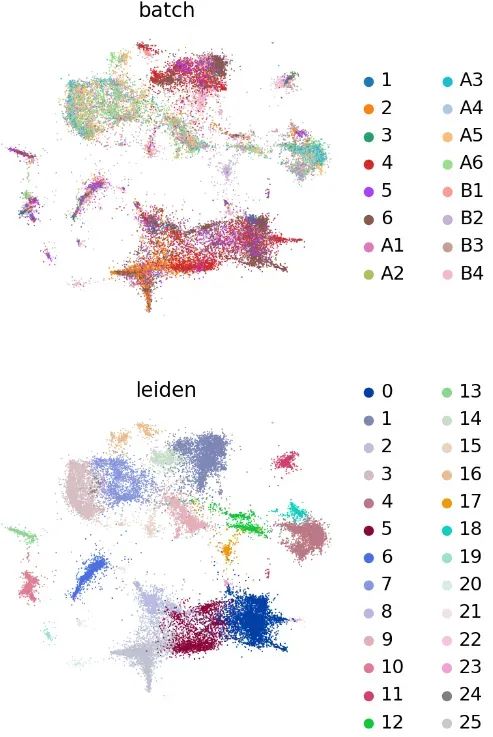
image-20230723214135712
Поскольку набор данных был аннотирован, эффект scVI можно оценить на основе результатов аннотации:
sc.pl.embedding(adata, basis="X_mde", color=["cell_type"], frameon=False, ncols=1)
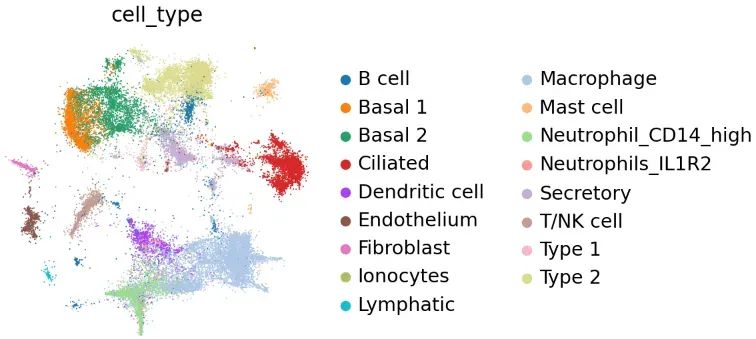
image-20230723214212112
Шаг 4. Используйте scANVI для интеграции.
Авторы утверждают, что использование scANVI может дать более точные результаты интеграции, если входные данные содержат предварительно аннотированную информацию.
Поскольку вышеизложенное обучило модель scVI, мы будем использовать ее для инициализации scANVI. При инициализации scANVI мы предоставляем ему labels_key. Поскольку scANVI также можно использовать для наборов данных с частично наблюдаемыми аннотациями, нам необходимо присвоить ему имя класса, соответствующее немаркированным ячейкам. Поскольку у нас нет непомеченных ячеек, мы можем дать ей любое случайное имя, не являющееся именем существующего типа ячеек.
lvae = scvi.model.SCANVI.from_scvi_model(
vae,
adata=adata,
labels_key="cell_type",
unlabeled_category="Unknown",
)
lvae.train(max_epochs=20, n_samples_per_label=100)
Добавить результаты как"X_scANVI"
adata.obsm["X_scANVI"] = lvae.get_latent_representation(adata)
Визуализация результатов:
adata.obsm["X_mde_scanvi"] = mde(adata.obsm["X_scANVI"])
sc.pl.embedding(
adata, basis="X_mde_scanvi", color=["cell_type"], ncols=1, frameon=False
)
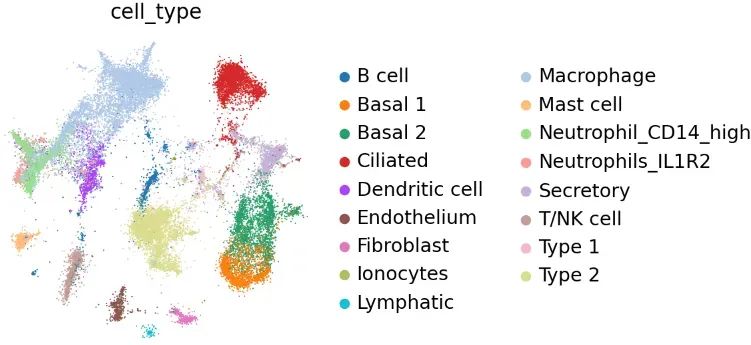
image-20230723215034705
Шаг 5. Рассчитайте эффект интеграции.
Здесь мы используем пакет scIB-metrics, который содержит масштабируемые реализации метрик, используемых в наборе тестов scIB. Мы можем использовать эти метрики для оценки качества ансамбля.
Мы видим, что дополнительное обучение с использованием информации на этикетках и scANVI улучшает показатели, отражающие биологическую сохранность (cLISI, этикетки Silhouette), не жертвуя при этом слишком большой возможностью коррекции партии (iLISI, партии Silhouette).
bm = Benchmarker(
adata,
batch_key="batch",
label_key="cell_type",
embedding_obsm_keys=["X_pca", "X_scVI", "X_scANVI"],
n_jobs=-1,
)
bm.benchmark()
bm.plot_results_table(min_max_scale=False)
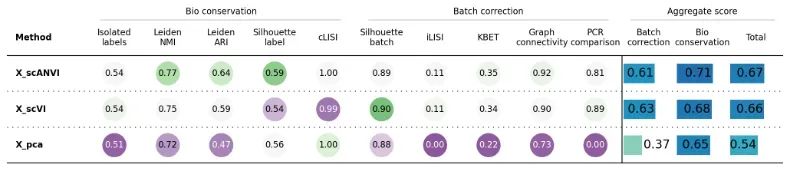
image-20230723215710408
df = bm.get_results(min_max_scale=False)
print(df)
Isolated labels Leiden NMI Leiden ARI \
Embedding
X_pca 0.50606 0.724169 0.474548
X_scVI 0.540021 0.750638 0.58713
X_scANVI 0.536124 0.771481 0.639793
Metric Type Bio conservation Bio conservation Bio conservation
Silhouette label cLISI Silhouette batch \
Embedding
X_pca 0.557418 1.0 0.878176
X_scVI 0.535189 0.994187 0.900951
X_scANVI 0.588762 0.998546 0.88585
Metric Type Bio conservation Bio conservation Batch correction
iLISI KBET Graph connectivity \
Embedding
X_pca 0.003306 0.223596 0.728504
X_scVI 0.111625 0.339272 0.899708
X_scANVI 0.105794 0.349628 0.922797
Metric Type Batch correction Batch correction Batch correction
PCR comparison Batch correction Bio conservation \
Embedding
X_pca 0.0 0.366716 0.652439
X_scVI 0.891112 0.628534 0.681433
X_scANVI 0.809051 0.614624 0.706941
Metric Type Batch correction Aggregate score Aggregate score
Total
Embedding
X_pca 0.53815
X_scVI 0.660273
X_scANVI 0.670014
Metric Type Aggregate score
3. Оценка и резюме
Я использовал этот алгоритм для запуска примеров данных, а также попробовал его на собственных данных. Моя первая мысль после его использования заключалась в том, что, когда я разбогатею в будущем, мне нужно будет купить высокопроизводительный сервер с графическим процессором. В настоящее время я беден и не могу позволить себе благородный алгоритм scvi-инструментов (чистая работа процессора очень медленная и очень тревожная).
Кроме того, scVI теперь можно запускать на языке R, о котором будет рассказано в следующем твите.
- END -
RasaGpt — платформа чат-ботов на основе Rasa и LLM.

Nomic Embed: воспроизводимая модель внедрения SOTA с открытым исходным кодом.

Улучшение YOLOv8: EMA основана на эффективном многомасштабном внимании, основанном на межпространственном обучении, и эффект лучше, чем у ECA, CBAM и CA. Малые цели имеют очевидные преимущества | ICASSP2023
Урок 1 серии Libtorch: Тензорная библиотека Silky C++

Руководство по локальному развертыванию Stable Diffusion: подробные шаги и анализ распространенных проблем
Полностью автоматический инструмент для работы с видео в один клик: VideoLingo

Улучшения оптимизации RT-DETR: облегченные улучшения магистрали | Support Paddle облегченный rtdetr-r18, rtdetr-r34, rtdetr-r50, rtdet
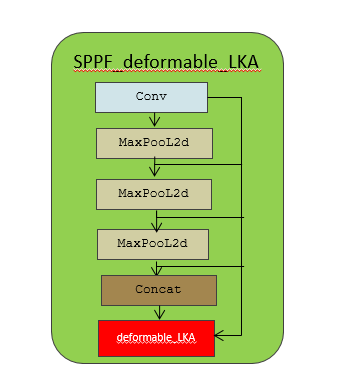
Эксклюзивное оригинальное улучшение YOLOv8: собственная разработка SPPF | Деформируемое внимание с большим ядром (D-LKA Attention), большое ядро свертки улучшает механизм внимания восприимчивых полей с различными функциями

Создано Datawhale: выпущено «Руководство по тонкой настройке развертывания большой модели GLM-4»!
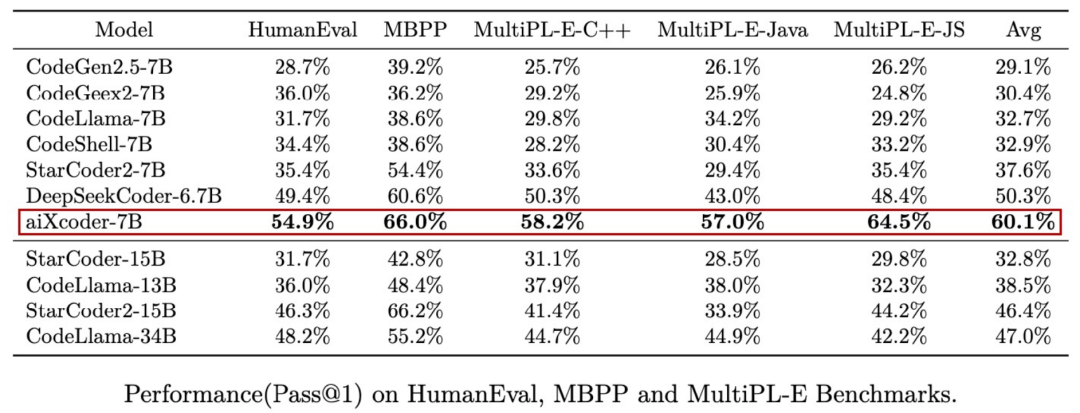
7B превышает десятки миллиардов, aiXcoder-7B с открытым исходным кодом Пекинского университета — это самая мощная модель большого кода, лучший выбор для корпоративного развертывания.
Используйте модель Huggingface, чтобы заменить интерфейс внедрения OpenAI в китайской среде.

Оригинальные улучшения YOLOv8: несколько новых улучшений | Сохранение исходной информации — алгоритм отделяемой по глубине свертки (MDSConv) |
Второй пилот облачной разработки | Быстро поиграйте со средствами разработки на базе искусственного интеллекта
Бесшовная интеграция, мгновенный интеллект [1]: платформа больших моделей Dify-LLM, интеграция с нулевым кодированием и встраивание в сторонние системы, более 42 тысяч звезд, чтобы стать свидетелями эксклюзивных интеллектуальных решений.

Решенная Ошибка | Загрузка PyTorch медленная: TimeoutError: [Errno 110] При загрузке факела истекло время ожидания — Cat Head Tiger

Brother OCR, библиотека с открытым исходным кодом для Python, которая распознает коды проверки.

Новейшее подробное руководство по загрузке и использованию последней демонстрационной версии набора данных COCO.

Выпущен отчет о крупной модели финансовой отрасли за 2023 год | Полный текст включен в загрузку |
Обычные компьютеры также могут работать с большими моделями, и вы можете получить личного помощника с искусственным интеллектом за три шага | Руководство для начинающих по локальному развертыванию LLaMA-3
Одной статьи достаточно для анализа фактора транскрипции SCENIC на Python (4)
Бросая вызов ограничениям производительности небольших видеокарт, он научит вас запускать большие модели глубокого обучения с ограниченными ресурсами, а также предоставит полное руководство по оценке и эффективному использованию памяти графического процессора!

Команда Fudan NLP опубликовала 80-страничный обзор крупномасштабных модельных агентов, в котором в одной статье представлен обзор текущего состояния и будущего агентов ИИ.

[Эксклюзив] Вы должны знать о новой функции JetBrains 2024.1 «Полнострочное завершение кода», чтобы решить вашу путаницу!

Краткое изложение базовых знаний о регистрации изображений 1.0

Новейшее подробное руководство по установке и использованию библиотеки cv2 (OpenCV, opencv-python) в Python.
Легко создайте локальную базу знаний для крупных моделей на основе Ollama+AnythingLLM.
[Решено] ошибка установки conda. Среда решения: не удалось выполнить первоначальное зависание решения. Повторная попытка с помощью файла (графическое руководство).

Одна статья поможет вам понять RAG (Retrival Enhanced Generation) | Введение в концепцию и теорию + практику работы с кодом (включая исходный код).
Эволюция архитектуры шлюза облачной разработки

