Фактический бой с одной ячейкой (1) Создание объекта загрузки данных, чтения данных и просмотра.
Основная структура статьи:
1. Загрузка данных
2. Чтение данных и создание объекта проверки.
- Чтение данных в различных форматах в условиях одиночного образца,Создание объекта seurat после чтения
- Несколько Чтение данных в различных форматах в условиях образцов, создание и объединение отдельных объектов после чтения.
1. Загрузка данных
Вы можете зайти на официальный сайт GEO (https://www.ncbi.nlm.nih.gov/gds), чтобы найти и загрузить нужные вам данные секвенирования отдельных клеток. Данные для тестирования примера кода будут предоставлены позже в этой статье.
Общие форматы данных секвенирования отдельных клеток, представленные в базе данных GEO, в основном включают следующее:
- 10x «matrix.mtx», «genes.tsv» и «barcodes.tsv» 10 раз Стандартный формат файла для данных секвенирования одноклеточного транскриптома Genomics. Эти файлы обычно хранятся в каталоге,Можно использоватьRead10Xфункция отRязык Читать в。
- Matrix.mtx: это файл разреженной матрицы, содержащий информацию об экспрессии генов в каждой отдельной клетке. Каждая строка матрицы представляет ген, каждый столбец представляет одну ячейку, а каждый элемент матрицы представляет уровень экспрессии гена в отдельной клетке.
- Genes.tsv (или Features.tsv): это текстовый файл, содержащий информацию о каждом гене. Каждая строка представляет ген, а каждый столбец представляет атрибут, например имя гена, номер гена и т. д.
- barcodes.tsv: это текстовый файл, содержащий информацию о штрих-коде для каждой отдельной ячейки. Каждая строка представляет одну ячейку, а каждый столбец представляет атрибут, например последовательность штрих-кода, тип ячейки и т. д.
- формат h5: Это двоичный формат файла, используемый для хранения крупномасштабных данных, которые могут содержать несколько типов данных, таких как матрицы, таблицы, изображения и т. д.
- Сжатая текстовая матрица (файл GZ в формате TXT или CSV): Сжатые текстовые матрицы можно использовать для хранения матриц выражений или метаданных для данных секвенирования одной ячейки, что может уменьшить размер файла и время передачи. 。
- формат h5ad: Он специально разработан для хранения и обмена данными об выражениях одной ячейки и использует библиотеку Anndata для создания и чтения. Формат h5ad совместим с такими инструментами, как cellxgene или Seurat, для визуализации и анализа данных отдельных клеток. 。
- h5seurat формат: Это формат файла, основанный на формате h5, который специально разработан для хранения и анализа мультимодальных экспериментов по экспрессии одной ячейки и пространственного разрешения, таких как CITE-seq или таких технологий, как 10X Visium. Формат h5seurat совместим с такими инструментами, как SeuratDisk, для чтения и записи одноячеечных данных.
- Файлы данных R (файлы RDS/RDATA): Матрица выражений хранится в формате файла данных Rязык и должна быть прочитана непосредственно программным обеспечением R.
2. Чтение данных и создание объекта проверки.
одиночный образец
одиночный образец чтения данных случая и демонстрация создания объекта seurat для каждого формата:
Формат 10x Genomics:
Загрузка демонстрационных данных:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE234527
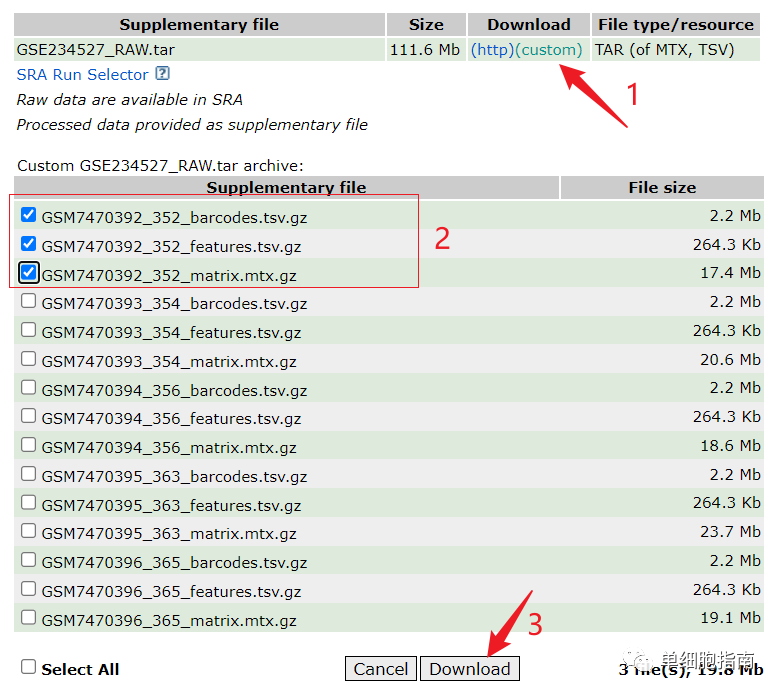
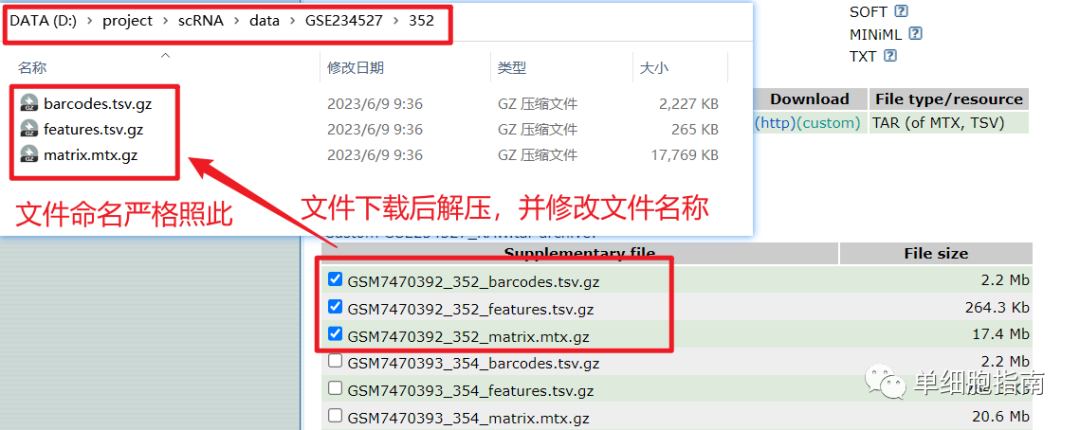
После скачивания файла разархивируйте его и измените имя. Путь к хранилищу следующий:
Ссылка на код для чтения файлов и создания объектов:
# Импортировать пакет Seurat
library(Seurat)
# Просмотр текущего рабочего каталога
getwd()
# Установить рабочий каталог (переключить рабочий каталог на указанный путь)
setwd("D:/project/scRNA")
# Чтение данных 10x, параметр data.dir указывает путь для хранения файла.
seurat_data <- Read10X(data.dir = "./data/GSE234527/352")
# Создать объект Сёра
seurat_obj <- CreateSeuratObject(counts = seurat_data,
project = "GSM7470392_352",
min.features = 200,
min.cells = 3)
# Просмотр основной информации об объектах Сера
seurat_objформат h5:
Загрузка демонстрационных данных:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE200874
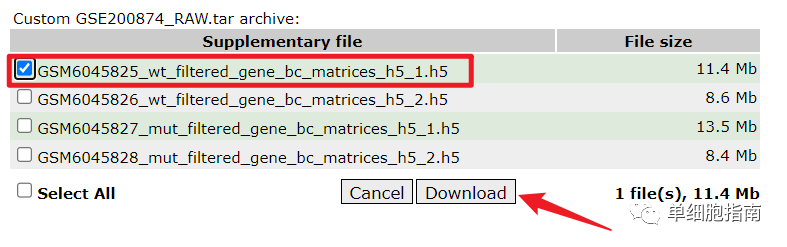
После скачивания разархивируйте его, путь хранения такой, как показано на рисунке.
Ссылка на код для чтения файлов и создания объектов:
# Импортировать пакет Seurat
library(Seurat)
# Просмотр текущего рабочего каталога
getwd()
# Установить рабочий каталог (переключить рабочий каталог на указанный путь)
setwd("D:/project/scRNA")
# Укажите местоположение и имя файла, который нужно прочитать.
h5_file <- "./data/GSE200874/GSM6045825_wt_filtered_gene_bc_matrices_h5_1.h5"
# Чтение файлов в формате h5 (используйте функцию Read10X_h5 для чтения файлов данных с одной ячейкой в формате h5)
seurat_data <- Read10X_h5(file = h5_file)
# Создать объект Сёра (с помощью функции CreateSeuratObject Создать объект Сёра,И преобразуем прочитанные данные формата h5 в объект Seurat)
seurat_obj <- CreateSeuratObject(counts = seurat_data,
project = "GSM6045825_wt",
min.features = 200,
min.cells = 3)
# Просмотр основной информации об объектах Сера
seurat_objСжатая текстовая матрица (файл GZ в формате TXT или CSV):
Рекомендуется вручную сжать эти два файла на локальный компьютер, чтобы проверить формат содержимого файла.
CSVсжатиеGZФормат Загрузка демонстрационных данных:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=gse130148
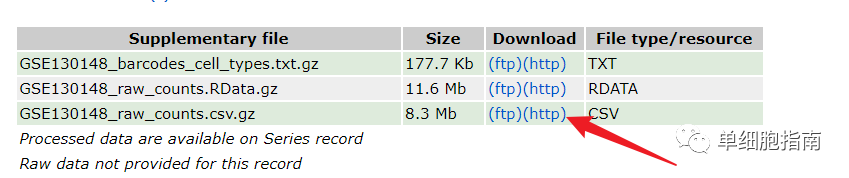
Путь хранения загруженного файла
Пример кода сжатия CSV в формате GZ:
# Импортировать пакет Seurat
library(Seurat)
# Просмотр текущего рабочего каталога
getwd()
# Установить рабочий каталог (переключить рабочий каталог на указанный путь)
setwd("D:/project/scRNA")
# Используйте функцию read.csv() для чтения данных из файла в формате csv.gz и используйте первый столбец в качестве имени строки.
seurat_data<- read.csv(gzfile("./data/GSE130148/GSE130148_raw_counts.csv.gz"), row.names = 1)
# Используйте функцию CreateSeuratObject() Создать объект Сёра,и укажите здесь название проекта
seurat_obj <- CreateSeuratObject(counts = seurat_data,
min.features = 200,
min.cells = 3,
project = "GSE130148")Пример кода сжатия txt в формате GZ:
# Импортировать пакет Seurat
library(Seurat)
# Просмотр текущего рабочего каталога
getwd()
# Установить рабочий каталог (переключить рабочий каталог на указанный путь)
setwd("D:/project/scRNA")
# Используйте функцию read.table() для чтения данных из файла в формате txt.gz и используйте первый столбец в качестве имени строки.
seurat_data<- read.table(gzfile("./data/GSE130xxx/xxxx.txt.gz"), row.names = 1, header = TRUE, sep = "\t")
# Используйте функцию CreateSeuratObject() Создать объект Сёра,и укажите здесь название проекта
seurat_obj <- CreateSeuratObject(counts = seurat_data,
min.features = 200,
min.cells = 3,
project = "GSE130xxx")
формат h5ad:
Скачать тестовые файлы:
https://www.dropbox.com/s/ngs3p8n2i8y33hj/pbmc3k.h5ad?dl=0
# Скачать тестовые файлы
# https://www.dropbox.com/s/ngs3p8n2i8y33hj/pbmc3k.h5ad?dl=0
# Импортируйте необходимые пакеты R.
library(Seurat)
# Установите пакет SeuratDisk
#remotes::install_github("mojaveazure/seurat-disk")
library(SeuratDisk)
# Просмотр текущего рабочего каталога
getwd()
# Установить рабочий каталог (переключить рабочий каталог на указанный путь)
setwd("D:/project/scRNA")
# Преобразуйте файл формата h5ad в файл формата h5seurat и укажите анализ, который будет использоваться как «РНК».
Convert("./data/pbmc/pbmc3k.h5ad", "h5seurat", overwrite = TRUE, assay = "RNA")
# использоватьLoadH5Seurat()функциянагрузкаh5seuratФорматдокумент,и Создать объект Сёра
seurat_pbmc <- LoadH5Seurat("./data/pbmc/pbmc3k.h5seurat")Файлы данных R (файлы RDS/RDATA)
# Используйте функцию load() для чтения файлов RDATA.
load("path/to/your/file.Rdata")
# Используйте функцию readRDS() для чтения файлов RDS.
my_data <- readRDS("path/to/your/file.rds")Несколько образцов
Несколько образцы корпуса, наша основная задача на10x Формат геномики и сжатая текстовая матрица (файл GZ в формате TXT или CSV)
10x формат Genomics. Несколько образцов чтения и создания объектов.:
Загрузка тестовых данных:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE234527
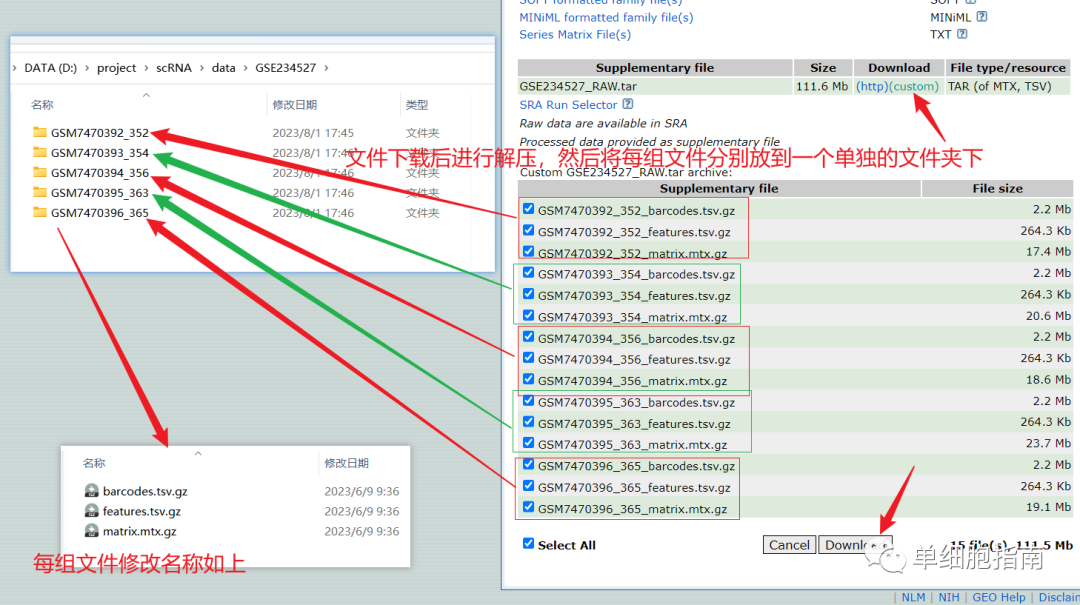
Пример кода:
# Импортировать пакет Seurat
library(Seurat)
# Просмотр текущего рабочего каталога
getwd()
# Установить рабочий каталог (переключить рабочий каталог на указанный путь)
setwd("D:/project/scRNA")
# Получить список всех файлов образцов в папке данных.
samples <- list.files("./data/GSE234527")
# Создайте пустой список для хранения объектов Сёра.
seurat_list <- list()
# Прочтите каждый образец10xданныеи Создать объект Сёра
for (sample in samples) {
# Путь к файлу сращивания
data.path <- paste0("./data/GSE234527/", sample)
# Чтение данных 10x, параметр data.dir указывает путь для хранения файла.
seurat_data <- Read10X(data.dir = data.path)
# Создать объект Сёра,и指定项目名称为样本документ名
seurat_obj <- CreateSeuratObject(counts = seurat_data,
project = sample,
min.features = 200,
min.cells = 3)
# Добавить объект Сёра в список
seurat_list <- append(seurat_list, seurat_obj)
}
# Распечатать список всех объектов Сера
seurat_list
# Объединить объекты Сёра, объединить все объекты Сёра в один объект
seurat_combined <- merge(seurat_list[[1]],
y = seurat_list[-1],
add.cell.ids = samples)
# Распечатайте объединенный объект Сёра
print(seurat_combined)Формат h5. Несколько образцов чтения данных и создания объектов:
Загрузка тестовых данных:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE200874
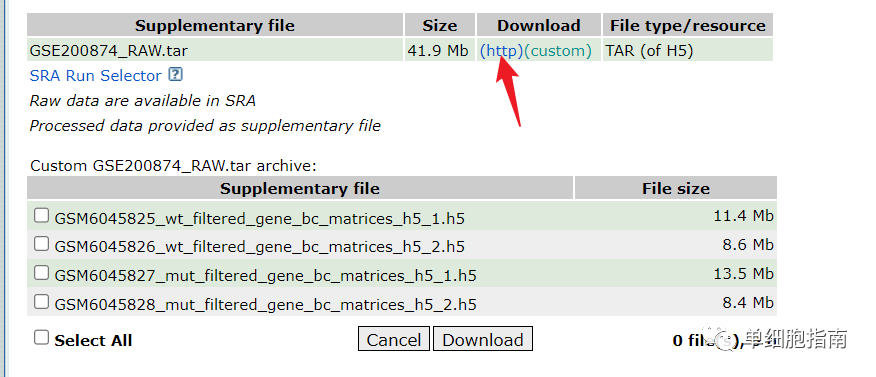
После скачивания разархивируйте данные:
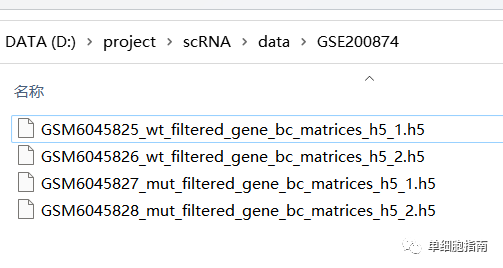
# Импортировать пакет Seurat
library(Seurat)
# Установить рабочий каталог
setwd("D:/project/scRNA")
# Получите список всех файлов h5 в папке данных.
h5_files <- list.files("./data/GSE200874", pattern = "\\.h5$")
# Создайте пустой список для хранения объектов Сёра.
seurat_list <- list()
# Перебрать каждыйh5документ的данныеи Создать объект Сёра
for (h5_file in h5_files) {
# Путь к файлу сращивания
data.path <- paste0("./data/GSE200874/", h5_file)
# Чтение данных h5
seurat_data <- Read10X_h5(filename = data.path)
# Создать объект Сёра,и指定项目名称为документ名
sample_name <- tools::file_path_sans_ext(basename(h5_file))
seurat_obj <- CreateSeuratObject(counts = seurat_data,
project = sample_name,
min.features = 200,
min.cells = 3)
# Добавить объект Сёра в список
seurat_list <- append(seurat_list, seurat_obj)
}
# Извлеките часть перед подчеркиванием
sample_names <- sub("_.*", "", h5_files)
# Объединить объекты Сёра, объединить все объекты Сёра в один объект
seurat_combined <- merge(seurat_list[[1]],
y = seurat_list[-1],
add.cell.ids = sample_names)
# Распечатайте объединенный объект Сёра
print(seurat_combined)Матрица сжатого текста (файл GZ в формате TXT или CSV).
Скачать тестовые файлы:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi
Загрузите три данных для демонстрации кода.
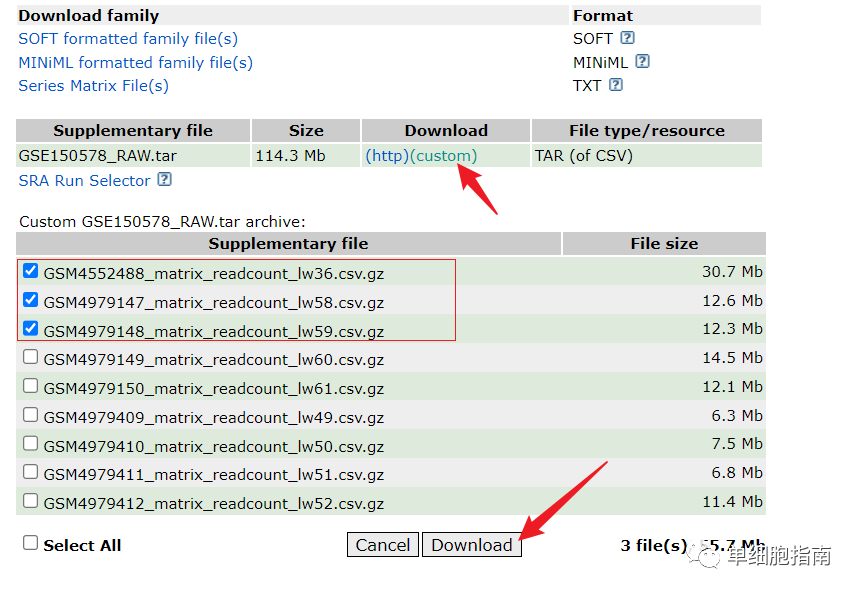
Разархивируйте после скачивания:
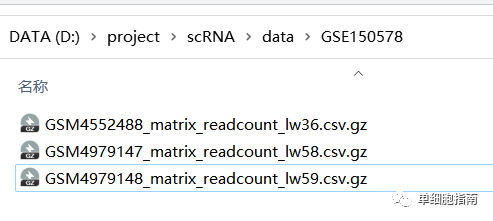
Пример кода:
# Импортировать пакет Seurat
library(Seurat)
# Импортировать пакет Seurat
library(Seurat)
# Установить рабочий каталог — это путь для хранения файла данных
setwd("D:/project/scRNA")
# Получить список всех файлов csv.gz
file_list <- list.files("./data/GSE150578", pattern = "\\.csv\\.gz$")
# Создайте пустой список для хранения объектов Сёра.
seurat_list <- list()
# Перебрать каждыйcsv.gzдокумент的данныеи Создать объект Сёра
for (file in file_list) {
# Путь к файлу сращивания
data.path <- paste0("./data/GSE150578/", file)
# Чтение данных файла csv.gz
seurat_data <- read.csv(gzfile(data.path), row.names = 1)
# Создать объект Сёра,и指定项目名称为документ名(удалить суффикс)
sample_name <- tools::file_path_sans_ext(basename(file))
seurat_obj <- CreateSeuratObject(counts = seurat_data,
project = sample_name,
min.features = 200,
min.cells = 3)
# Добавить объект Сёра в список
seurat_list <- append(seurat_list, seurat_obj)
}
# Извлеките часть перед подчеркиванием
sample_names <- sub("_.*", "", file_list)
# Объединить объекты Сёра, объединить все объекты Сёра в один объект
seurat_combined <- merge(seurat_list[[1]],
y = seurat_list[-1],
add.cell.ids = sample_names)
# Распечатайте объединенный объект Сёра
print(seurat_combined)Вы можете использовать более эффективные функции, такие как fread(), чтобы заменить функцию read.csv() в этой статье, но вам следует обратить внимание на то, является ли формат данных после чтения точным.
Справочная ссылка: https://www.jianshu.com/p/5b26d7bc37b7.
Ссылка на ссылку: https://mp.weixin.qq.com/s/M15kWdH8eDONfakNhY-enA

Углубленный анализ переполнения памяти CUDA: OutOfMemoryError: CUDA не хватает памяти. Попыталась выделить 3,21 Ги Б (GPU 0; всего 8,00 Ги Б).
[Решено] ошибка установки conda. Среда решения: не удалось выполнить первоначальное зависание. Повторная попытка с помощью файла (графическое руководство).
Прочитайте нейросетевую модель Трансформера в одной статье

.ART Теплые зимние предложения уже открыты

Сравнительная таблица описания кодов ошибок Amap

Уведомление о последних правилах Points Mall в декабре 2022 года.

Даже новички могут быстро приступить к работе с легким сервером приложений.
Взгляд на RSAC 2024|Защита конфиденциальности в эпоху больших моделей
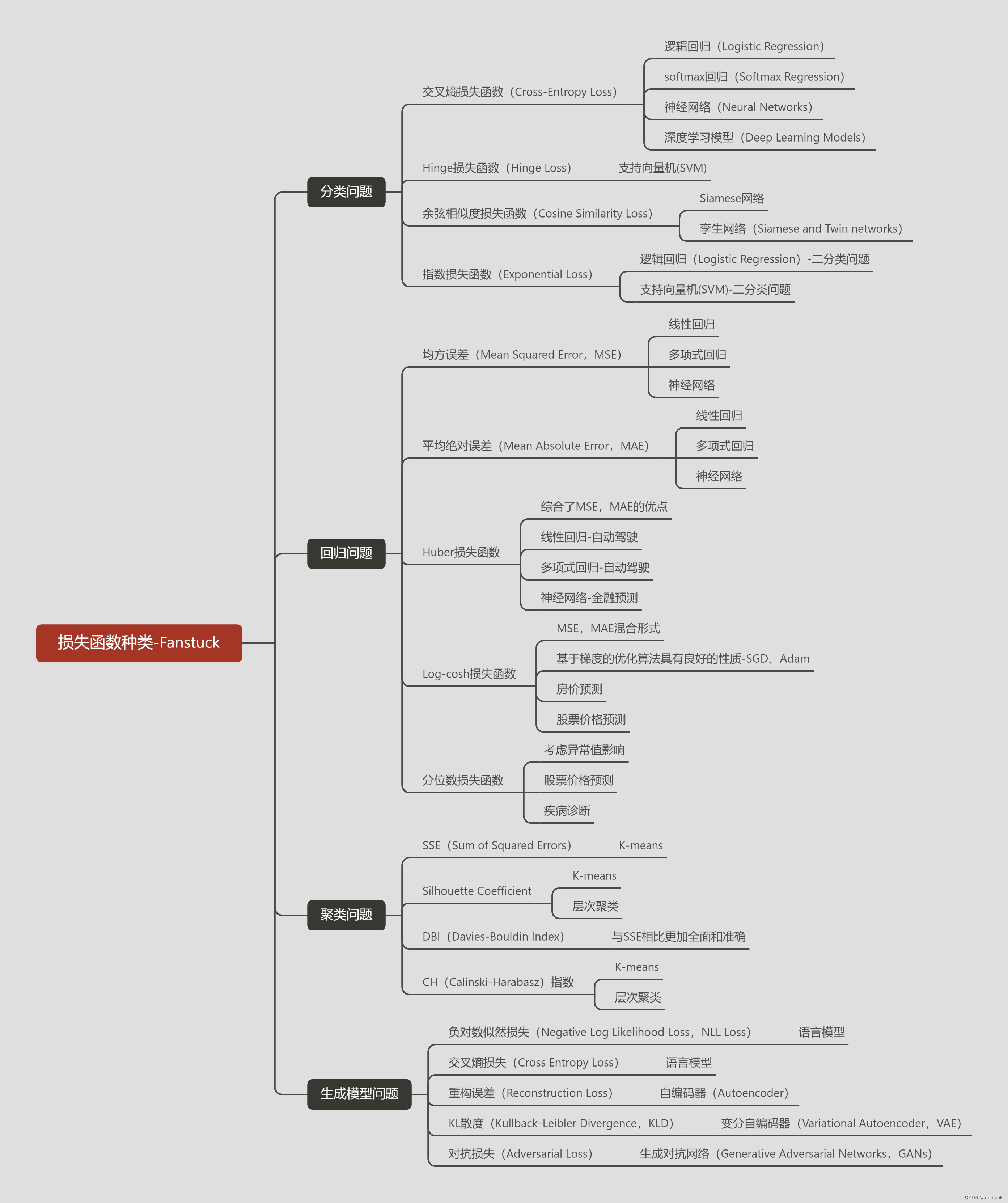
Вы используете ИИ каждый день и до сих пор не знаете, как ИИ дает обратную связь? Одна статья для понимания реализации в коде Python общих функций потерь генеративных моделей + анализ принципов расчета.

Используйте (внутренний) почтовый ящик для образовательных учреждений, чтобы использовать Microsoft Family Bucket (1T дискового пространства на одном диске и версию Office 365 для образовательных учреждений)
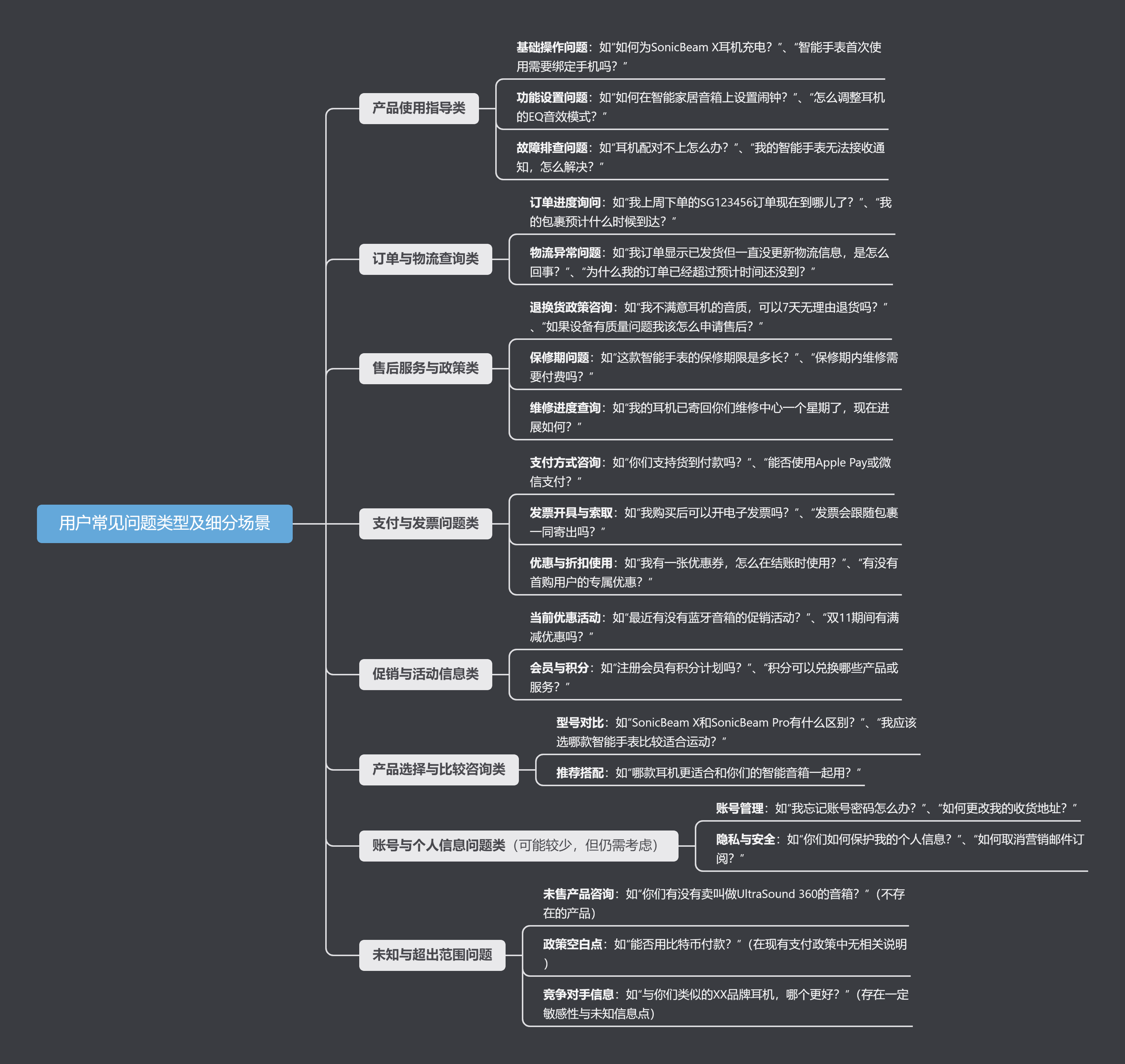
Руководство по началу работы с оперативным проектом (7) Практическое сочетание оперативного письма — оперативного письма на основе интеллектуальной системы вопросов и ответов службы поддержки клиентов

[docker] Версия сервера «Чтение 3» — создайте свою собственную программу чтения веб-текста

Обзор Cloud-init и этапы создания в рамках PVE
Корпоративные пользователи используют пакет регистрационных ресурсов для регистрации ICP для веб-сайта и активации оплаты WeChat H5 (с кодом платежного узла версии API V3)

Подробное объяснение таких показателей производительности с высоким уровнем параллелизма, как QPS, TPS, RT и пропускная способность.
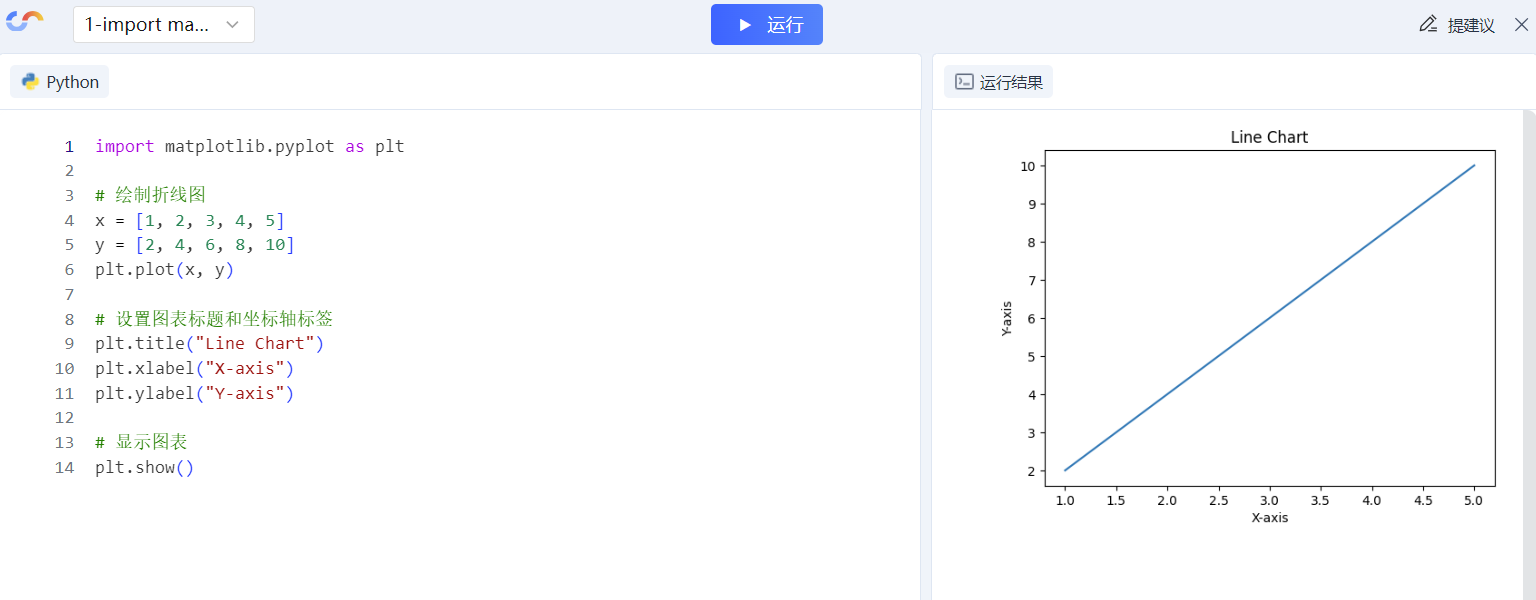
Удачи в конкурсе Python Essay Challenge, станьте первым, кто испытает новую функцию сообщества [Запускать блоки кода онлайн] и выиграйте множество изысканных подарков!

[Техническая посадка травы] Кровавая рвота и отделка позволяют вам необычным образом ощипывать гусиные перья! Не распространяйте информацию! ! !

[Официальное ограниченное по времени мероприятие] Сейчас ноябрь, напишите и получите приз

Прочтите это в одной статье: Учебник для няни по созданию сервера Huanshou Parlu на базе CVM-сервера.

Cloud Native | Что такое CRD (настраиваемые определения ресурсов) в K8s?

Как использовать Cloudflare CDN для настройки узла (CF самостоятельно выбирает IP) Гонконг, Китай/Азия узел/сводка и рекомендации внутреннего высокоскоростного IP-сегмента

Дополнительные правила вознаграждения амбассадоров акции в марте 2023 г.
Можно ли открыть частный сервер Phantom Beast Palu одним щелчком мыши? Супер простой урок для начинающих! (Прилагается метод обновления сервера)
[Играйте с Phantom Beast Palu] Обновите игровой сервер Phantom Beast Pallu одним щелчком мыши

Maotouhu делится: последний доступный внутри страны адрес склада исходного образа Docker 2024 года (обновлено 1 декабря)

Кодирование Base64 в MultipartFile
5 точек расширения SpringBoot, супер практично!

Глубокое понимание сопоставления индексов Elasticsearch.